Your browser does not fully support modern features. Please upgrade for a smoother experience.
Please note this is an old version of this entry, which may differ significantly from the current revision.
Subjects:
Biochemistry & Molecular Biology
癌症是全球第二大死因,并且是一种遗传性疾病,具有高度遗传性。全基因组关联研究 (GWAS) 已经确定了许多风险相关位点,其中包含数百个单核苷酸多态性 (SNP)。其中一些与癌症相关的 SNP 已被揭示为因果关系,并且在某些情况下阐明了癌症风险关联机制的功能特征。
- genome-wide association analysis
- single nucleotide polymorphism
- cancer
- molecular and biological mechanism
1. 编码区 SNP 的功能机制
位于编码区的SNP可分为两类:同义突变和非同义突变。虽然同义突变不影响蛋白质的氨基酸序列,但它们可能通过影响转录后修饰、翻译率和其他过程来改变蛋白质的表达。相反,非同义单核苷酸多态性(nsSNPs)引起氨基酸的取代,从而导致蛋白质结构、物理和化学性质(稳定性、溶解性等)及其功能发生变化。目前,有许多生物学软件包(如 SIFT(Sorting Intolerant From Tolerant)、F-SNP(功能单核苷酸多态性)和 PolyPhen)可用于预测 nsSNPs 对蛋白质结构和功能的影响 [ 41 , 42, 43 ]。与位于基因非编码区的 SNP 相比,肿瘤相关 nsSNP 的功能机制相对简单 [ 44 ]。结合全外显子分析,已确定多个编码区 SNP 与结直肠癌的发展相关。例如,位于 SH2B3 结构域的错义突变 rs3184504 (p. trp263ARg) 可能会在调节细胞分裂的背景下改变蛋白质的功能。其他编码变体也可能影响可变剪切(RS16888728、UTP23)[ 45 ]。位于基因编码区内的 SNP 影响疾病风险的机制与所产生的编码蛋白的功能密不可分。
一些风险位点对产生的蛋白质的氨基酸序列产生影响。示例包括肺癌 [ 46 ] 和乳腺癌 [ 47 ]中的 BRCA2 p.Lys3326Ter (rs11571833) 和 CHEK2 p.Ile157Thr (rs17879961) 。假定对此类变体的机械解释相对简单。除了上述之外,编码 SNP 还会影响 RNA 加工;一个例子是 TP53 的 3' 非翻译区中的 rs78378222,风险校正变异将序列 AATAAA 更改为 AATACA,从而改变 TP53 的聚腺苷酸化信号,并最终导致 TP53 mRNA 的 3'-末端加工受损 [ 48 , 49]. 一些变异也会影响剪接。Tian 和他的同事在ELP2基因中发现了一个单核苷酸变异,它通过剪接数量性状基因座 (sQTL)影响ELP2外显子前体 mRNA 剪接 [ 50 ]。
研究人员通常专注于感兴趣的特定信号通路、基因和基因修饰,同时还进行全外显子关联分析 (GWAS) 以发现对这些分子和过程有重大影响的任何相关编码 SNP。例如,Li 及其同事使用外显子测序并对参与 TGF-β 信号传导的 12 个重要基因进行了关联分析,发现 TGF-β 通路中的低频致病变异有助于结直肠癌 (CRC) 易感性。他们发现位于 SMAD7 基因中的错义变异 rs3764482 (c. 83C>T; p. S28F )始终与 CRC 风险密切相关。与显性等位基因 C 相比,rs3764482 等位基因 T 在限制 TGF-β 信号传导和通过阻碍下游基因的激活减少受体调节的 SMADs (R-SMADs) 的磷酸化方面更有效,从而促进癌细胞增殖并促进 CRC发病机制 [ 51 ]。
编码 SNP 也可能影响基因和蛋白质修饰。N6-甲基腺苷 (m6A) 修饰对于确保信使 RNA 的稳定性至关重要,并参与许多生物活动,包括前 mRNA 剪接、3' 端加工、核输出、翻译调节、mRNA 降解和 DNA 损伤反应 [ 52 , 53 ]。m6A 甲基化修饰发生在信使 RNA (mRNA) 中,可由甲基化“写入器”形成,并由去甲基化“擦除器”去除 [ 54 ]。位于 ANKLE1 基因中的Rs8100241被鉴定为与 CRC 和乳腺癌的易感性相关。rs8100241 风险等位基因 A 的存在(图 1a) 与 m6A“编写器”复合体(由蛋白质 METTL3、METTL14 和 WTAP 组成)和 m6A“读取器”蛋白 (YTHDF1) 结合,被发现可增加基因ANKLE1上的 m6A 修饰水平,从而增加其蛋白表达。从机制上讲,ANKLE1 通过减少 CRC 细胞增殖同时保持基因组完整性,作为潜在的肿瘤抑制因子发挥作用,从而有助于降低 CRC 的风险 [ 55 ]。
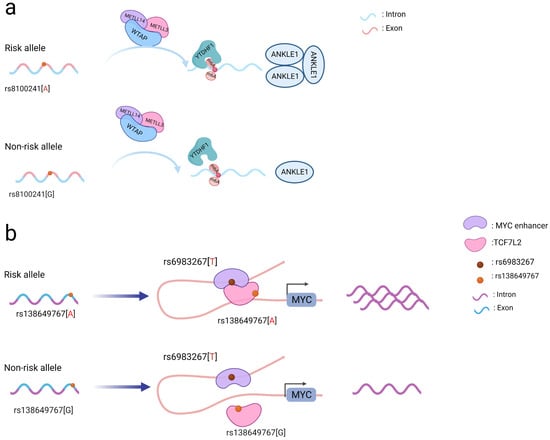
图 1. SNP 编码所采用的作用机制示意图。( a ) 在ANKLE1第二外显子区域发现的 rs8100241 变体的 A 等位基因通过增加ANKLE1 mRNA m6A 水平并因此促进 ANKLE1 蛋白表达而与较低的 CRC 风险相关联,从而可能起到负调节剂的作用通过维持基因组稳定性来阻碍细胞生长。( b ) MYC增强子和启动子中TCF7L2错义变体 rs138649767 和调节变体 rs6983267之间的相互作用对 MYC表达的影响。
值得注意的是,编码 SNP 可能与其他 SNP 相互作用以产生更强的功能作用 [ 45 ]。位于TCF7L2外显子区的rs138649767 A等位基因(图1b)可激活含有rs6983267等位基因G的MYC增强子,促进MYC的表达[ 56 ]。SMAD7外显子和内含子中出现的SNP可能会影响其调控,并共同影响涉及 SMAD7 和 TGFβ 的下游信号通路 [ 51 ]。因此,在检查编码区中的 SNP 时,应考虑它们之间的相互作用,以更好地了解它们的功能过程。
2. 非编码区 SNP 的功能机制
越来越多的证据表明,非编码区的 SNP 是人类基因组中最常见的遗传变异类型,占个体间变异的 90% [ 6 , 57 ]。根据位置的不同,该区域可以包含近端(启动子、增强子或超增强子)或远端(基因间或基因内)的反应元件。GWAS鉴定的风险位点位于细胞类型特异性活性染色质的基因组区域,其中大部分是数量性状位点、甲基化数量性状位点和转录因子(TF)结合相关位点。染色质构象研究有助于将 SNP 定位的调控区域与其各自的靶基因联系起来 [ 6 , 58 , 59]. 这些位点可能参与基因转录、转录后加工、翻译、翻译后修饰和其他调节基因表达的过程。已经使用表达数量性状位点 (eQTL) 鉴定了许多靶基因,以检测 SNP 与基因表达之间的关系。非编码 SNP 可以通过序列近端 ( cis ) 或远端 ( trans ) 调节靶基因的转录)-相互作用。研究发现,此类风险 SNP 区域的组蛋白修饰特别丰富,尤其是与启动子和增强子活性相关的区域(H3K4me3、H3K4me1、H3K27ac)。预计大多数 SNP 会破坏特定转录因子的结合基序。例如,rs6983267 可能会改变 MYC、CTCF 和 TCF7L2 等转录因子的结合 [ 16 ]。除了通过改变转录因子结合位点 (TFBS) 影响基因转录水平外,非编码 SNP 还会改变表观遗传修饰和/或染色质结构以影响靶基因表达。通过上述方法,非编码SNP参与细胞增殖、凋亡、迁移和侵袭。
2.1. 改变启动子的遗传变异
启动子是一段 DNA 序列,它被识别、结合并用于启动 RNA 聚合酶的转录。启动子包含 RNA 聚合酶特异性结合和转录起始所需的保守序列的变异。大多数启动子位于结构基因转录起始点的上游,启动子本身不被转录[ 60 ]。启动子位于给定结构基因 5' 端的上游,它们激活 RNA 聚合酶以准确结合模板 DNA,特异性地诱导转录启动 [ 60 ]]. 启动子本身不控制基因活动;相反,基因活性是通过与称为转录因子 (TF) 的蛋白质结合来调节的。启动子区域内的 SNP 通常通过影响此类转录因子的结合发挥调节作用。最近报道的一个例子是位于死亡受体 4 (DR4) 启动子的 SNP rs13278062,它会改变结直肠癌的风险。研究表明,rs13278062 G>T 变异改变了转录因子 Sp1/NF1 的结合亲和力,增加了 DR4 的表达,从而抑制了结直肠癌的发生和转移 [ 61 ]。MPO 启动子 SNP rs2333227 通过改变启动子对 AP-2α 的亲和力来增加结直肠癌的恶性特征 [ 62]. 位于 MSMB 基因上游启动子的变异 SNP rs10993994 也被发现在前列腺癌患者中过度表达;这归因于更强的 CREB 结合,从而增加了启动子活性 [ 63 ]。此外,SNP rs11672691 是与前列腺癌相关的风险基因座,与 lncRNA PCAT19 相关。非风险变体 rs11672691 及其连锁不平衡 (LD) SNP rs887391 更有可能将转录因子 NKX3.1 和 YY1 与 PCAT19-short 启动子结合,从而导致启动子增加但增强子活性降低,然后激活 PCAT19-short , 并最终导致较低的前列腺癌易感性 [ 64]. GWAS 在乳腺癌中发现了多个基因启动子区域的 SNP,包括 TERT、KLHDC7A、PIDD1 和 ESR1,报告者研究表明独立的风险等位基因会改变目标启动子活性[ 65、66 ]。大多数报告的启动子变化通过改变 TF 结合发挥其调节作用。SNP rs3824662 等位基因 A(图2a )通过改变 TF GATA3 表达增加染色质可及性,促进 GATA3 与 CRLF 启动子的结合,并最终形成染色质环 [ 67 ]。
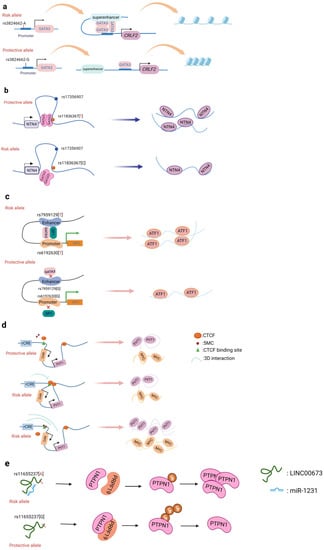
图 2.非编码 SNP 使用的作用机制示意图。( a ) SNP rs3824662 等位基因 A 通过诱导 GATA3 表达、促进 GATA3 与 CRLF 启动子的结合并最终形成染色质环来增加染色质可及性。( b ) NTN4 增强子风险变体 rs11836367 与 TF GATA3 结合以调节 NTN4 表达,最终促进乳腺癌的发生和发展。( c ) 增强子 SNP rs7959129 风险等位基因 G 与启动子 SNP rs6192603 风险等位基因 G 相互作用,通过结合 TF GATA3 和 SP1 促进 ATF1 表达。( d) 风险等位基因 rs11986220 和 –10 Kb 处更高的甲基化协同作用赋予更高的肿瘤风险;然而,当-20 Kb 被低甲基化时,风险 SNP 的功能被 CTCF 介导的增强子阻断绝缘子环抑制。( e ) LINC00673 中的风险变异 rs11655237 创建了一个 miR-1231 结合位点,该位点会干扰 LINC00673 的表达并导致胰腺癌易感性。
2.2. 改变增强子的遗传变异
增强子是 DNA 序列的区域,可以增加其靶基因序列的顺式作用转录。每个增强子与其目标启动子的距离不同;在哺乳动物物种中,增强子可以距其目标基因 100 bp 到 Mb [ 68]. 与启动子不同,增强子可以在基因的任何地方找到;它们可以位于目标基因的上游或下游,甚至位于另一个基因的基因体内,增强子调控可以绕过其他基因,而不管它们的方向如何。增强子必须与特定的蛋白质因子结合以增强其目标的转录。增强子通常具有组织或细胞特异性,因此它们仅在某些细胞或组织中表现出活性,这取决于这些细胞或组织中存在的特定蛋白质因子 [ 69 ]。增强子通常由表观遗传标记 H3K4me1 和 H3K27ac 识别,它们存在于活性增强子元件中。相反,H3K27me3 被认为是与较低增强子活性相关的沉默表观遗传标记 [ 70, 71 ]。GWAS 确定的常见疾病风险位点通常出现在非编码区域,其中许多被认为起到增强剂的作用 [ 72 ]。根据新出现的数据,这些 SNP 可能通过改变重要转录因子与关键转录增强子的结合来影响基因调控 [ 73 ]。
2.2.1. 乳腺癌
在所有癌症中,迄今为止发现的风险位点数量最多的是乳腺癌 [ 13 ]。了解恶性转化的驱动机制提供了对抗癌症复发和治疗耐药性的前景。张等。确定 SNP rs4971059 位于第六个内含子和 TRIM46 基因的活性增强子元件内。通过使用 CRISPR/Cas9 介导的同源重组,他们构建了等位基因 G 转换为等位基因 A 的 SNP rs4971059,从而导致 TRIM46 过表达,促进乳腺癌细胞生长,增强体外化疗耐药性,并加速体内肿瘤发展 [ 74 ] ]. 此外,Yang 及其同事(图 2b) 报告了 NTN4 位点 (12q22) 的非编码调控变异 rs11836367,并将其确定为与乳腺癌风险相关的因果变异。rs11837367 保护性 T 等位基因促进 GATA3 与远端增强子结合并增加 NTN4 表达 [ 75 ]。
2.2.2. 前列腺癌
几项研究已经独立鉴定了特定前列腺癌 (PCa) 易感基因座中的几个基因,这些基因要么由含有顺式调控元件 (CRE) 的致病 SNP 控制,要么被指定为 SNP 相关基因 [ 76 ]。6q22 的 SNP rs339331 被发现是一种前列腺癌风险相关变异。已发现 rs339331 的风险等位基因 T 可增强 HOXB13 的增强子结合,以等位基因特异性方式改变 RFX6 蛋白的水平,并赋予前列腺癌易感性 [ 77 ]。最近,Huang 等人。还发现位于增强子元件内的 PCa 相关 rs11672691 可以改变 HOXA2 的结合位点,从而通过影响附近基因的表达促进肿瘤发生 [78 ]。
值得注意的是,还有其他 SNP 案例导致不同转录因子中的 DNA 结合多态性。例如,已发现位于lncPSCA内含子增强子中的胃癌风险相关多态性 (rs2978980 T>G) 会破坏转录因子 RORA 的结合,从而以等位基因特异性方式导致较低的lncPSCA表达[ 79 ]。作为另一个例子,已发现 rs2647046 增强子与HLA-DQB1-AS1启动子相互作用,以等位基因特异性方式通过 CTCF 介导的长程环改变其表达,从而赋予对肝细胞癌 (HCC) 的易感性 [ 80]. 远处基因间区域染色体 11q13.3 的另一个变异已被表征为肾细胞癌的易感位点。为了控制转录,11q13.3 基因座编码了一个与CCDN1启动子物理连接的远程增强子 [ 81 ]。有趣的是,SNP 位点可以同时充当启动子和增强子,它们的转换由背景基因型决定。因此,一个基因可以产生几种不同的 RNA,这些 RNA 与疾病的发展有关。SNP rs11672691 介导不同基因型下的启动子和增强子转换。PCAT19长增强子中的风险相关序列与PCAT19相互作用-long 启动子通过激活细胞周期基因来促进前列腺癌的发展 [ 64 ]。
2.2.3. 大肠癌
GWAS 已经确定了许多结直肠癌风险基因座,但只有这些基因座的一小部分靶基因被系统地询问过。例如,Yu 等人。在基因CDH1的内含子中鉴定了一个常见的 SNP (rs7198799) 。他们证明 rs7198799 的风险等位基因 C 作为增强子可以靶向 TF NFATC2 并远程增强 ZFP90 表达 [ 82]. SNP 变体影响细胞特异性增强子功能的一个重要机制是通过改变 TF 结合,从而调节靶基因的表达。田等。确定了分别位于 ATF1 启动子和第一个内含子中的两个风险 SNP(rs61926301 和 rs79591129)。这些富含增强子区域和开放染色质,它们也与 H3K4me1、H3K27ac 和 ATAC-seq 峰相关。这两种变体通过优先结合两个 TF SP1 和 GATA3 来增加 ATF1 的表达 [ 83 ]。Rs174575 可以作为 FADS2 和lncRNA-AP002754.2的特异性远程增强子在转录因子 E2F1 的参与下。有趣的是,TF E2F1可促进FADS2的表达,形成染色质环,影响结直肠癌的发生[ 84 ]。
2.3. 影响启动子-增强子相互作用的遗传变异
启动子-增强子相互作用 (PEI) 是差异转录调控的基础。几种技术(染色体构象捕获 (3C)、Hi-c 和H3K27Ac - HiCHIP)允许研究远程顺式调节[ 85、86、87 ] 。启动子-增强子相互作用是当前转录控制理论中涉及的基本事件。到目前为止,几乎没有证据表明增强子靶基因的转录控制需要 PEI。启动子的插入或缺失、某些 PEI 相关蛋白的缺失以及 PEI 破坏绝缘子的包含都会对靶基因的表达产生影响。田等。发现两个风险变体(rs1926301 和 rs7959129)位于ATF1启动子和内含子,分别;前者结合 TF SP1,而后者结合 TF GATA3(图2c)。他们发现这两个风险位点通过结合 SP1 和 GATA3、促进 ATF1 表达并赋予 CRC 遗传易感性来增加启动子和增强子之间的相互作用 [ 83 ]。此外,SNP rs11672691 以依赖于不同背景基因型的方式介导启动子和增强子转换。风险由 PCAT19-long 增强子与 PCAT19-long 启动子相互作用决定,从而通过激活细胞周期基因改变前列腺癌的发展 [ 64 ]。
2.4. 改变 3D 基因组结构的遗传变异
在细胞核内,基因组 DNA 折叠成一个三维结构,通过形成染色质环在不同层次组织起来。这些结构可以将远距离增强子带到它们的目标启动子附近,从而影响基因表达和调控。染色体折叠成染色质,其特征是序列调节空间相互作用,这是维持正常细胞状态和功能的关键。在癌症基因组中,结构变异通常会导致基因组 3D 结构发生变化,从而导致基因组介导的转录控制发生改变 [ 88 ]。三维基因组结构或高级染色质结构的变化与多种疾病的发生和发展有关 [ 89 , 90]. 长距离染色质环主动或被动调节癌症易感基因。增强子经常与其靶基因启动子区域形成远程染色质环以影响基因表达。例如,9q22 基因座包含与甲状腺癌风险相关的 SNP rs965513,它划定了与 PTC 风险密切相关的 33-kb 连锁不平衡块(包括先导 SNP rs965513)。该块的染色质特征和调控元件特征表明至少三个调控元件作为增强子起作用。使用染色体构象捕获技术,研究人员在人类乳头状甲状腺癌细胞系 (KTC-1) 和未受影响的甲状腺组织中观察到这些元件与 FOXE1 和 PTCSC2 共有的启动子区域的长程环状连接 [91 ]。同样,张等人。发现 rs1859962 风险相关 LD 块包含一个 PCa 特异性增强子,它与SOX9基因形成一个 1-Mb 染色质环。这项研究发现,rs1859962 PCa 风险 LD 阻断通过连接到 E1 增强子的长距离染色质环接触SOX9 [ 92 ]。
CTCF 是一种转录因子,可通过环化促进远程染色体接触。霍夫曼等人。发现 7 号染色体上 Igf2/H19 印记控制区 (ICR) 中的一个等位基因与 11 号染色体上 Wsb1/Nf1 的一个等位基因共定位。CTCF 的缺乏或母体 ICR 的消融被发现消除了这种联系并改变了Wsb1/Nf1基因的表达[ 93 ]。这一发现证实了 CTCF 在控制染色质形状和由此产生的基因表达方面的重要性。另一方面,CTCF 的独特贡献在于绝缘体。绝缘子是短的核苷酸序列,它决定了彼此靠近的基因组区域的边界 [ 94 ]]. 当 CTCF 与绝缘体区域结合时,它会通过干扰增强子和基因启动子之间的通讯来抑制基因转录 [ 95 ]。艾哈迈德 M. 等人。通过执行 CRISPRi 筛选识别(图2d)非编码顺式调控元件 (rCRE)。他们发现 8q24.21 区域广泛标记有 H3K27ac,并且与 AR、FOXA1 和 HOXB13 具有显着的结合亲和力,所有这些都是 PCa 发病机制的重要转录调节因子 [ 96 ]。使用涉及 ChIP、Hi-C、CRISPR 和功能拯救的综合方法,研究人员还发现包含 rCRE 序列的 rs11986220 与MYC相互作用启动子在 V16A 细胞中而不是在 22Rv1 细胞中,因为启动子与 CRE 的相互作用通常由上游 10 kb 区域的 CTCF 位点促进,从而防止染色质环化 [ 96 ]。同样,rs6702619 区域居住着 CTCF,它充当绝缘体,与 CRC 相关位点进行远程物理相互作用 [ 97 ]。了解 CTCF 介导的 3D 基因组结构将有助于了解 CTCF 位点或调控增强子位点的非编码 GWAS SNP 的潜在作用机制 [ 98 ]。
2.5. 影响 miRNA 结合的遗传变异
微小 RNA (miRNA) 是非编码 RNA 分子,通过调节信使 RNA 降解和翻译影响基因表达。MicroRNA 通常被 RNase iii 酶 Dicer 从 60-110 个核苷酸长的发夹前体(折叠)RNA 结构(pre-miRNA)中切除,然后整合到 RNA 诱导的沉默复合物(RISC)中。pro-miRNA 序列由 Pol-II [ 99 ] 转录。越来越多的证据表明,miRNA 通过与目标 mRNA 的 3'-UTR 结合,在致癌作用中发挥关键作用 [ 100 ]。MiRNA 突变或其错误表达与人类恶性肿瘤和癌症相关基因表达的改变有关 [ 101 ]。霍夫曼等人。在has-miR-196a-2中检测到一个变体 (rs11614913)使用 GWAS 筛选 15 种 miRNA 中的遗传变异。该 SNP 被确定与降低的乳腺癌风险相关 [ 102 ]。先前的研究已经证实,miRNA区域[ 103 ]岛的甲基化可能改变miRNA的功能,从而影响致癌途径。作者和他的同事发现,miRNA 前体上游区域的 CpG 岛与乳腺癌风险相关 [ 102 ]。ATF1 rs11169571变体通过影响hsa-miR-1283和hsa-miR-520d-5p结合显示与 ATF1 表达密切相关,这可能会增加结直肠癌的易感性 [ 56 ]]. 此外,位于 MDM4、CD44、LAMC1 和其他基因的3'UTR区域的SNP发挥类似的机制[ 104、105、106 ]。
长链非编码 RNA 中的一些 SNP 也可以改变它们与 miRNA 的结合亲和力。在 lncRNA RP11-362K14.5 (CCSlnc362)的外显子 1 处发现的变异基因座 rs1317082为miR-4658建立了一个结合位点,从而降低了 CCSlnc362 的表达并降低了对 CRC 的易感性 [ 107 ]。lncRNA LOC146880 中的rs140618127与非小细胞肺癌之间的联系涉及miR-539-5p结合位点。已发现miR-539-5p和LOC146880的组合导致致癌基因ENO1的激活减少。减少ENO1磷酸化还导致较低的 PI3K 和 Akt 激活,这与细胞增殖和肿瘤形成减少有关 [ 108 ]。此外,LINC00673 外显子中的 SNP rs11655237 等位基因 G 可以创建一个 miRNA 结合位点,从而增加 LINC00667 表达的功能(图2e)。此外,RCC [ 109 ] 中的 rs67311347、CRC [ 110 ] 中的 rs12982687 和颈部鳞状细胞癌 (HNSCC) 中的 rs16854802 [ 111 ] 是 lncRNA 序列中的 SNP,它们通过与 miRNA 结合影响靶基因表达。如果一个 SNP 出现在 miRNA 中,它会因此影响 miRNA 与靶基因的结合亲和力。
This entry is adapted from the peer-reviewed paper 10.3390/cancers14225636
This entry is offline, you can click here to edit this entry!
