L'aAutofagia è un processo di degradazionephagy is a highly conserved cellulare altamente conservato che regola il degradation process that regulates cellular metabolismo cellulare e l' and homeostasi in condizionis under normali e fisiopatologiche. L'autofagia e il and pathophysiological conditions. Autophagy and metabolismo sono collegati nel sistema are linked in the hematopoietico, svolgendo un ruolo fondamentale nell'auto-rinnovamento, sopravvivenza e system, playing a critical role in the self-renewal, survival and differenziazione delle cellule staminali e progenitrici tiation of hematopoietiche e nella morte cellulare, influenzando in stem and progenitor cells, and in cell death, particolare il destino cellulare del pool di cellule staminali ularly influencing the cell fate of the hematopoietiche. Nella leuc stem cell pool. In leukemia, l'autofagia sostiene la crescita delle cellule leucemicheautophagy supports leukemia cell growth, contribuisce alla sopravvivenza delle cellule staminali leucemiche e allates to leukemia stem cell survival and resistenza alla chemioterapia. La leucemia mieloide acutance to chemotherapy. Acute myeloid leukemia (AML), un tipo comune di leucemia acuta con scarsa sopravvivenza e prognosi, è una malattia ematopoietica clonale che colpisce le cellule staminali e progenitrici ematopoietiche, che provoca il blocco della differenziazione mieloide e la soppressione delle funzioni ematopoietiche.a common type of acute leukemia with poor survival and prognosis,
1. Autofphagia e cellule y and AML Stem Cellstaminali AML
Dur
ain
te lo sviluppo leucemico, le LSC possono adattare il lorog leukemic development, LSCs can adapt their metabolism
o e i meccanismi autofagici per fornire l'elevata and autophagic mechanisms to provide the high energ
ia e i y and nutrient
i necessari per las required for LSC prolifera
zione e la sopravvivenza delle LSC in condizioni di carenza di nutrienti, fame, ipossia o durante i trattamenti chemioterapicition and survival under conditions of nutrient deficiency, starvation, hypoxia, or during chemotherapy treatments [7
, 91
, 92
, 93
, 94
] .
La mMito
fagia, la funzione mitocphagy, mitochondrial
e e l' function and integrit
à possono influenzare la vitalità, la y can affect the viability, prolifera
zione e il potenziale dition and differen
ziazione e latiation potential and longevit
à delley of normal
i cellule staminali ema hematopoietic
he (HSC stem cells (HSCs) [95]
eand sonoare important
i nella in the survival strateg
iay of diAML s
opravvivenza delle cellule staminali AML (LSC)tem cells (LSCs) .
In Sp
articolare, la proteina chinasi (AMPK) attivata dall'adenosina5′-monofosfato (AMP), uecifically, adenosine 5′-monophosphate (AMP)-activated protein kinase (AMPK), a protein comple
sso proteico fondamentale nel metabolismo mitocx critical in mitochondrial
e e nella mitofagia, è co metabolism and mitophagy, is constitutiv
amente attivata nelle LSC, aumentando la clearanceely activated in LSCs, increasing mitoc
hondrial
e per clearance to support
are la crescita e la sopravvivenza delle LSC attraverso il suo obiettivo a valle LSC growth and survival through its downstream target FIS1,
la fissionthe mitoc
hondrial
e 1 componente fission 1 protei
co di un comp
lessoonent of a mitoc
hondrial
e che promuove la fissione complex that promotes mitoc
hondrial
e fission [
96 ].
LAMPK/FIS1-media
mitofagia mediata da AMPK/FIS1 è necessaria per l'auto-rinnovamento e la sopravvivenza delle LSC ted mitophagy is required for LSC self-renewal and survival [
96].
Una soOv
raeserexpression
e di of FIS1
è stata trovata anche nelle cellule AML mentre lawas also found in AML cells while deple
zione dition of FIS1
compromette la mitofagia, indebolendo laimpairs mitophagy, weakening the self-renewal capacit
à di auto-rinnovamento delle LSC e determinando l'induzione dellay of LSCs and resulting in the induction of myeloid differen
ziazione mieloide mediante l'inattivazione ditiation by inactivation of GSK3 (gl
icogeno sintasi chinasi 3), indicando così la mitofagia come meccanismo di regolazione per laycogen synthase kinase 3), thus pointing to mitophagy as a regulatory mechanism for AML progression
e della LMA [
96 ].
PiùMore recent
emente, la perdita del ly, the loss of sequestosom
ae 1 (
SQSTM1 ),
also kno
to anche come p62, un recettorwn as p62, a selective auto
fagico selettivophagy receptor crucial
e per lo sviluppo e la for the development and progression
e dell' of AML in vivo, induc
e l'es the accumul
o diation of damaged mitoc
ondri danneggiati e hondria and mitochondrial supero
ssido mitocondriale,xide, thereby comprom
ettendo così la sopravvivenza delle cellule leucemiche. Quindi, la perditaising cell survival. leukemia cells. Thus, the loss of di SQSTM1 coimp
romette lairs leukemia progression
e della leucemia nei in mouse model
li murini di LMA, sottolineando il ruolo della mitofagia nella sopravvivenza delles of AML, underscoring the role of mitophagy in LSC survival. LSC.97 ]. Co
mplle
ssivamente, questictively, these studi
dimostrano che è necessaria una maggiore attività autofagica delle LSC per laes demonstrate that increased autophagic activity of LSCs is required for malignant progression
e maligna in into AML.
TuttaHowev
ia,er, in contra
riamente all'attivazione dell'autofagia osservata nell'AML, una perdita di autofagia nelle HSC sane innesca l'esst to the activation of autophagy observed in AML, a loss of autophagy in healthy HSCs triggers the expansion
e di una popolazione di cellule of a population of progenit
rici nel midollo osseo, dando origine a una mior cells in the bone marrow, resulting in severe and invasive myeloprolifera
zione grave e invasiva, come nell'AML umana [tion, as in human AML [ 53
] .
QueThis
to apparent
e parado
sso può essere spiegato dai ruolix may be explained by the distin
ti che l'autofagia può svolgere durante lact roles that autophagy may play during AML progression
e dell'AML, che può essere diversa nelle varie fasi della leuc, which may be different at various stages of leukemogenesi
s [
98 ,
99].
L'aAuto
fagia nelle HSC normali può prevenire l'insorgenza del cancro, come meccanismo sophagy in normal HSCs may prevent the onset of cancer, as a tumor suppressiv
o del tumore. Infatti, l'autofagia rimuove glie mechanism. Indeed, autophagy removes damaged organell
i danneggiati, come ies, such as mitoc
ondri, e protegge le cellule hondria, and protects hematopoieti
che dall'c cells from genomic instabilit
à genomica e dall'infiammazione,y and inflammation, thus preven
endo così l'insorgenza della leucting the onset of leukemia.
In particolare, l'aumento del danno al DNA, gli alti livelli di ROS, l'Notably, increased DNA damage, high ROS levels, aneuploid
ia e uny, and an aberrant accumul
o aberrante diation of p62/SQSTM1
sono stati have been correlat
i con un processo autofagico alteratoed with an impaired autophagy process, indica
ndo un ruolo chiave dell'autofagia nellating a key role of autophagy in preven
zione dell'inizio delting the initiation of tumor
e [100
] .
Al cCon
trario, nel cancro accertato, l'autofagia può funzionare come un percorso favorevole che promuove la sopravvivenza e la crescita del tumoreversely, in established cancer, autophagy may function as a favorable pathway that promotes tumor survival and growth,
aiutand
So lme cellule tumorali a sfuggire allo stress metabolico e agli stimoli di morte.
Alcuni studies have also s
tudi h
anno anche dimostrato che l'attivazione del flusso autofagico gioca un ruolo solo nell'inizio dell'AML, con una traown that the activation of the autophagic flux only plays a role in the initiation of AML, with a transforma
zione da HSC ation from HSC to LSC,
e, quindi, dopo questa fase, l'autofagia non è necessaria per il mantenimento della malattia and, therefore, after this stage, autophagy is not required for disease maintenance [101
] .
GliAML studi
sull'AMLes MLL-AF9,
l'the most common altera
zione più comune nell'AML infantile, indicantion in childhood AML, point to ATG5 o
r ATG7
come necessari per l'insorgenza dell'AML, ma una volta stabilita la condizione leucemica, l'autofagia non è richiesta per la funzioneas required for the onset of AML, but once the leukemic condition is established, autophagy is not required for function LSC in vivo [101
, 102
] .
TuttaHowev
ia, in un diverso modello di topoer, in a different AML MLL-ENL
, il knockout mouse model, Atg5 o
r Atg7
ha ridotto il numero di LSC funzionali, aumentato l'attivazione mitocondriale e i livelli di ROS in queste cellule e prolungato la sopravvivenza dei topi lknockout reduced the number of functional LSCs, increased the
ucemici [103] . ]. In
quethis
to conte
sto, durante il xt, during the process
o di leuc of leukemogenesi
, la metilazione dell'istone può regolare gli effettori autofagici centrali e i regolatori autofagici a monte come s, histone methylation can regulate central autophagic effectors and upstream autophagic regulators such as ATG 5
eand ATG7
per influenzare indirettamente il livello di autofagia [to indirectly affect the level of autophagy [104 104] ].
Insieme, questiTogether, these studi
es sugge
riscono un ruolo altamente st a highly comple
sso e dix and context-dependent
e dal contesto per l'autofagia nella tra role for autophagy in leukemic transforma
zione leucemica rispetto alltion with respect to the maintenance prop
rietà di mantenimento delle LSC nell'erties of LSCs in AML.
IlThe du
plice ruolo dell'autofagia nell'AML, comeal role of autophagy in AML, as a promot
ore o soer or suppressor
e del cancro nell'AML, è ancora oggetto di dibattito. Gli studi hanno dimostrato che l'autofagia può agire come meccanismo pro o of cancer in AML, is still a matter of debate. Studies have demonstrated that autophagy can act as a pro- or anti-proliferativ
o a seconda del lignaggio e del contestoe mechanism depending on the lineage and molecular genot
ipico molecolare della malattia, riflettendo il grado di ypic context of the disease, reflecting the degree of heterogeneit
à dell'y of AML [105
] .
2. Regoulazione dei geni dell'autofagia nelle cellule AMLtion of autophagy genes in AML cells
Numerousi studi hanno dimostrato che l'aumento dell'autofagia nelle cellule AML es have demonstrated that increased autophagy in AML cells conferisces protezione dal trattamento chemioterapico e promuove la sopravvivenza delle ction from chemotherapy treatment and promotes AML cell survival.
Increllule AML.
L'auased ATG7-me
ntdiat
a autofagia mediata da ATG7 è stata ased autophagy has been associat
a a scarsi risultatied with poor clinic
i e a una breveal outcomes and short durat
a dellaion of remission
e nei pazienti con LMA in AML patients [
106 ].
PiùMore recent
emente, alcunly, some protein
e coinvolte nella sopravvivenza delle cellule leucemiche e sovraess involved in leukemia cell survival and overexpresse
nell'AML sono state cord in AML have been relate
alla sovraesd to ATG overexpression
e di ATG, alla base dell'interazione tra autofagia e sovraes, underlying the interplay between autophagy and protein overexpression
e proteica che promuove la sopravvivenza delle cellule leucemiche that promotes leukemia cell survival [8]
.]. Hu et al.
hanno d
imostrato che un'emonstrated that elevat
a esed expression
e di of SIRT1 (Sirtuin 1),
un attore chiave nella biogenesia key player in mitoc
hondrial
e e nella proteina correlata all'autofagia, è biogenesis and autophagy-related protein, is associat
a a un'ed with elevat
a esed expression
e di of CXCR4,
un markera negative prognostic
o negativo nell'AML e ad altre marker in AML, and other protein
e cors relate
all'autofagia comed to autophagy such as ATG5
eand LC3 in
campioni umani primari di AMLprimary human AML samples, indica
ndo un ruolo potenziale della via di segnalazioneting a potential role of the SDF-1α-CXCR4
nell'induzione dell'autofagia nelle cellule AML, che promuove ulteriormente la loro sopravvivenza sottosignaling pathway in the induction of autophagy in AML cells, which further promotes their survival under stress [107
] .
Il canalThe
ionico del recettore transitorio della transient melastatin
a receptor 2 (TRPM2)
, coinvolto nel mantenimento della sopravvivenza cellulare dopo il danno ossidativo, è altamente espresso nell' ion channel, involved in the maintenance of cell survival after oxidative damage, is highly expressed in AML [
108 ].
Eseguendo l'esaurimento dBy performing TRPM2 depletion , Chen SJ et al. [
108 ]
hanno d
imostrato che i livelli di proteine emonstrated that the levels of ULK1, Atg7
eand Atg5
sono diminuiti nelle cellule impoveriteproteins are decreased in di TRPM2- depleted cells ,
portando all'inibizione dell'autofagia. È ileading to inhibition of autophagy. Important
ely, sottolineareTRPM2 chde
l'esaurimento dipletion in TRPM2 nell'AML in
hibi
sce lts leukemia prolifera
zione della leucemia e aumenta la sensibilità alla tion and increases the doxorubicin
a delle cellule AML sensitivity of AML cells [
108 ].
StFu
di funzionali sunctional studies in normal
i cellule CD34 + CB hanno CD34+CB cells indicat
o che l'inibizione dell'esed that inhibition of VMP1 expression
e di VMP1 ha ridotto il flusso autofagico, con diminuzione dell'espansione delle cellule staminali e reduced autophagic flux, with decreased hematopoietic stem and progenit
rici ematopoieticheor cell (HSPC)
, d expansion, delayed differen
ziazione ritardata, aumento dell'tiation, increased apoptosi
e compromissione della funzione cellulare e dell'attecchimentos, and impaired cellular function and engraftment in vivo.
Risultati simili sono stati osservati nelle linee cellulari leucemiche e nelle cellule AMLSimilar results were observed in leukemic cell lines and primary CD34+
primarie. Inoltre, l'analisi AML cells. Furthermore, ultrastru
tturale hactural analysis indicat
o che le cellule leucemiche che sovraesprimonoed that leukemic cells overexpressing VMP1 ha
nno un numero ridotto di strutture mitocondriali e il numero di strutture di dve a reduced number of mitochondrial structures and the number of lysosomal degrada
zione lisosomiale è aumentato. La sovraestion structures is increased. Overexpression
e di of VMP1 (vacuole membrane protein-1)
ha aumentato il flusso autofagico e migliorato la qualità mitocondriale, che ha coinciso con un aumento della soglia per la perdita indotta da venetoclax della permeabilizzazione della membrana esterna mitocincreased autophagic flux and improved mitochondrial
e (MOMP) e dell'apoptosi nelle cellule leucemiche. quality,109 ].
DHe
lezioni eterozigoti, mutazioniterozygous deletions, missens
o o cambiamenti nel numero di copie dei geni chiave dell'autofagia sono stati trovati con un'alta e mutations, or changes in copy number of key autophagy genes have been found with a high frequen
za nei pazienti con LMA, in cy in AML patients, partic
olare nei pazienti con LMA con cariotipi complessiularly in AML patients with complex karyotypes [5
, 103
] . In partic
oular
e, una perdita c, a heterozygous chromosom
ica eterozigote di al loss of 5q, 16q
o 17p è , or 17p correlat
a allees with region
i che codificano i geni autofagicis encoding the autophagic genes ATG10 eand ATG12 ,
GABARAPL2 e, and MAP1LC3B o
r GABARAP , r
iespe
ttivamentectively [
103 ],
eand m
olti altri geni autofagici hanno un basso livello di esany other autophagic genes have a low level of expression
e nell'AML umano in the Human AML blast
i, un flusso autofagico ridotto e alti livelli dis, a reduced autophagic flux, and high levels of ROS [
103 ].
Inoltre, uno studio ha suggerito che i geni chiave dell'autofagiaFurthermore, one study suggested that key autophagy genes such as comeULK1 ,
ATG3 ,
ATG4D eand ATG5 wer
anoe significa
tivamente sottoregolati nelle cellule AML primarie rispetto aintly downregulated in primary AML cells compared to normal
i granuloc
itiytes [
110 ].
3. Biomarcatori aAutofagicphagic biomarkers
ReceSign
temente sono stati compiutiificant progress
i significativi per has recently been made to identif
icarey specific
i geni correlati all'autofagia per la previsione degli esiti autophagy-related genes for the prediction of clinic
i nell'AML. Insieme ai genial outcomes in AML. Along with the previously described ATG prge
cedentemente descritti, diversines , several microRNA
s implicat
i nella leuced in leukemogenesi
e nella chemis and chemoresist
enza sono stati coinvolti anche nell'attivazione dell'autofagia e possono essere utilizzati come ance have also been involved in autophagy activation and can be used as biomar
catorikers [
111 ].
In pNota
rticolare, la sovraesbly, miR-17-5p overexpression
e di miR-17-5p nella leuc in leukemia prom
uove laotes AML prolifera
zione di AML inibendo l'autofagia attraverso iltion by inhibiting autophagy through BECN1 targeting
BECN1 [
112 ,
113 ,
114 ]. Gansan et al. d
imostrato che le cellule emonstrated that stromal
i sottoregolano i livelli di cells downregulate miR-23a-5p
nelle cellule leucemiche, portando alla sovraregolazione dell'autofagialevels in leukemia cells, leading to upregulation of prote
ttiva in questctive autophagy in these cell
ule, aumentando così la loros, thereby increasing their resist
enza alla tossicità della chemioterapiaance to chemotherapy toxicity.
115 ].
È stato dimostrato chOve
la sovraesrexpression
e di of MiR-143
migliora lahas been shown to enhance the sensi
bilità delle cellule AML alla citotossicità del trattamento con citivity of AML cells to the cytotoxicity of cytarabin
ae (Ara-C)
inibendo l'autofagia attraverso il treatment by inhibiting autophagy through targeting ATG7
eand ATG2B [
116 116] ].
Una sovraesAn overexpression
e di of miR-15a-5p
è coinvolta nella chemiis involved in the chemoresist
enza dei pazienti con LMA, attraverso i geni correlati all'autofagiaance of AML patients, through the autophagy-related genes ATG9A ,
ATG14 ,
GABARAPL1 eand SMPD1 which
e prendono di mira le cellule AML target AML cells [
117 ].
I rRecent
i progressi nella advances in bioinformatic
a hanno prodotto una firma correlata all'autofagia che può aiutare a prevedere la sopravvivenza globales have yielded an autophagy-related signature that may help predict overall survival (OS)
e/o gli esiti and/or clinic
i dei pazienti con LMA. Diversial outcomes of patients with AML. Several studi
hanno dimostrato che la es have shown that the progression
e dell'AML dipende dalla firma genica of AML depends on the gene signature associat
a all'autofagiaed with autophagy [
118 ].
UnA recent
e studio di bioinformatic
a ha costruito un modello contenente 10 geni correlati all'autofagia per prevedere la sopravvivenza dei pazienti affetti da AML, dimostrando che i gruppi ad alto rischio di AML hanno una maggiore ess study constructed a model containing 10 autophagy-related genes to predict survival of AML patients, demonstrating that groups at high AML risk have higher expression
e di geni del of immune checkpoint
immunitario e una genes and a higher percent
uale più alta di cellule age of CD4 T
e NK and NK cells [119]
.].
InFurthermo
ltre, questo studio è stato in grado di predire l'OS nella LMA attraverso la firma dire, this study was able to predict OS in AML through the signature of 10 gen
i, indicando questoes, pointing to this model
lo come un efficace predittor as an effective prognostic
o per i pazienti con LMA, utile per guidare la predictor for AML patients, useful to guide patient stratifica
zione dei pazienti petion for immunot
herapie
e farmaci [119s and medications [119] ]. .
The L
o studio di ASSO Cox bioinformatic
as regression
e LASSO Cox che ha study which identifi
cato una firma di rischio critico peed a critical risk signature for AML, co
stituito dai geni autofagicinsisting of the autophagic genes BAG3 ,
CALCOCO2 ,
CAMKK2 ,
CANX ,
DAPK1 ,
P4HB ,
TSC2 eand ULK1 ,
ha
veva un eccd excellent
e potere predittivo per la predictive power for AML prognosi
AMLs [
120].
In pNota
rticolare, le citochine ibly, immunos
ouppressive
sono risultatcytokines were found to be significa
tivamente aumentate nel microambiente tumorale di pazienti con un alto rischio di ntly increased in the tumor microenvironment of patients with a high risk of AML, pre
visto sulla base della firma correlata all'autofagia di questi pazientidicted on the basis of the autophagy-related signature of these patients [120
] .
TuttaHowev
ia, il valorer, the prognostic
o della firma ATG in ambito value of the ATG signature in the clinic
o è ancora dibattuto. Pertanto, i ruoli della firma ATG e dell'autofagia nella patal setting is still debated. Therefore, the roles of the ATG signature and autophagy in the pathogenesi
dell'AML dovrebbero essere ulteriormente studiatis of AML should be further investigated.
InFurthermo
ltre, uno studio ire, an interes
sante ha ting study indicat
o che una firma died that an autophagy-related lncRNA
correlata all'autofagia contenente seisignature containing six lncRNA
s (
HYMAI ,
MIR155HG ,
MGC12916 ,
DIRC3 ,
C1orf220 eand HCP5 )
puòmay avere uhave an important
e valore prognostic
o value [
121 ].
UnoA studio recent
e ha indicato quattro lncRNA study pointed to four autophagy-associat
i all'autofagiaed lncRNAs (
MIR133A1HG ,
AL359715.1 ,
MIRLET7BHG , eand AL356752.1 )
comeas firma da utilizzaresignatures to poten
zialmente cometially be used as a biomar
catore per predire la sopravvivenza dei pazienti con LMA ker to predict survival of AML patients [
122 ].
Compllessivamente, questi datictively, these data indicano che il ruolo dell'autofagia nello sviluppo del tumore dipende chiaramente dal tipo di LMA e dallo stadio di sviluppo del tumore. Inoltre, l'autofagia può fornire alle cellule tumorali una te that the role of autophagy in tumor development clearly depends on the type of AML and stage of tumor development. Furthermore, autophagy may provide cancer cells with a survival strategia di sopravvivenzay, suggerendo un uso tsting a therapeutico per l'inibizione dell'autofagia. D'altra parte, l'autofagia può indurre la morte cellulare, indicando l'attivazione dell'autofagia come una nuova use for autophagy inhibition. On the other hand, autophagy can induce cell death, pointing to autophagy activation as a novel strategia nella terapia del cancro. Pertanto, èy in cancer therapy. Therefore, it is necessariy to determinare il ruolo svolto dall'autofagia nei sottotipie the role played by autophagy in the molecolari diular subtypes of AML, o il grado di sviluppo del tumore, perr the degree of tumor development, to verificare se la suay whether its modulazione potrebbe portare ation could lead to benefici per il paziente trattatots for the treated patient.
4. Autofphagia ey and genetic alterazioni genetiche nell'tions in AML
IlThe AML fenotipo AML deriva da molteplici alterazioniphenotype results from multiple molec
olariular, genetic
he e, and epigenetic
he che influenzano la alterations that affect the differen
ziazione, la tiation, prolifera
zione e l'tion, and apoptosi
deis of myeloid progenitor
i mieloidi. L's. The World Health Organiz
zazione Mondiale della Sanità haation has classifi
cato le AML in base allaed AMLs based on the presen
za dice of partic
olariular genetic altera
zioni genetiche, spesso otions, often originat
e da traslocazioni cromosomiche o altri riing from chromosomal translocations or other genome rearrang
iamenti del genoma come ements such as t(8;21), t(15;17), inv(16), inv
(3) , t(6;9), t(9;11) o
r t(11;19), o
mutazioni nelle chinasi del recettore, in mediatori di segnalazione chiaver mutations in receptor kinases, key signaling mediators, proto-oncogen
i o enzimi ees, or epigenetic
i, ad esempio enzymes, for example, muta
zionitions in FLT3 (FMS
- like t
iyrosin
a chinasie kinase 3),
TP53 ,
c-KIT o
r IDH1/2 ,
NPM1 (nucleo
fosmina 1) e proteina legante potenziatore CCAAT phosmin 1) and CCAAT enhancer binding protein (
CEBPA ) [
1 ,
2 ,
123 ].
QuThes
te muta
zioni nelletions in AML ha
nno uve an impa
tto sulla scelta della terapia più adattact on the choice of the most suitable therapy.
L'The associa
zione tra autofagia e alterazionition between autophagy and recurrent genetic
he ricorrenti è stata alterations has been descri
tta in diversibed in several AML studi
sull'AML, ma necessita di ulteriori indaginies, but needs further investigation [
124 ,
125 ].
QuiHere,
riassumiamo e aggiorniamo i we summarize and update recent
i progressi che hanno evidenziato il legame tra autofagia e geni di advances that have highlighted the link between autophagy and fusion
e e mutazioni genes and recurrent oncogenic
he ricorrenti nell'AML e il coinvolgimento dell'autofagia nel trattamento chemioterapico mutations in AML and the involvement of autophagy in chemotherapy treatment (
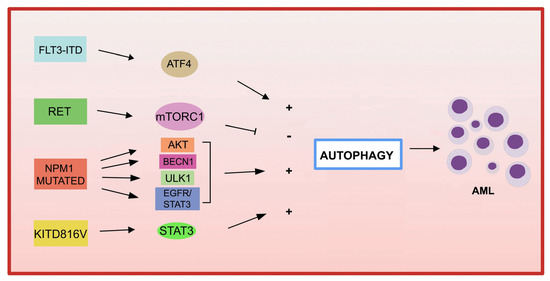
Figurae 1 ).
Figurae 1. MGene mutazioni genetiche nell'AML e nell'autofagia. Diverse anomalietions in AML and autophagy. Several recurrent genetiche ricorrenti nell'AML sono coinvolte nella abnormalities in AML are involved in the deregolazione dell'autofagia, portando allulation of autophagy, leading to leukemia progressione della leucemia.
4.1. Geni di fFusionen genes in AML e autofagiaand autophagy
La maggiMo
r parte dei casi di APL sono causati da una traslocazione de novo t st cases of APL are caused by a de novo translocation t(15;17)
(q22; (q22;q21),
che risulta nellaresulting in the fusion
e del gen of the
RARA cogen
il genee with the PML gene [
126 ,
127 ].
APL
e cellule APL che hanno un'es cells that have lower expression
e inferiore di geni correlati all'autofagia rispetto alle cellule normali hanno un'attività autofagica ridotta. Utilizzando agenti of autophagy-related genes than normal cells have reduced autophagic activity. differenzianti, come l'acido all Using differentiating agents, such as all- trans- retino
id acid
e (ATRA)
e il triossido di arsenicoand arsenic trioxide (ATO)
attualmente utilizzati in ambito currently used in clinic
o, il livello di esal settings, the expression
e dei geni correlati all'autofagia aumenta, ripristinando così l'autofagia nelle cellule APL level of autophagy-related genes increases, thus restoring autophagy in APL cells [
128].
EnBot
rambi gli agenti possono attivare l'h agents can activate ETosis,
un tipo di morte cellularea type of cell death mediat
a dal rilascio di trappoleed by the release of neutrophil extracellular
i di neutrofili traps (ET).
Inoltre, l'azione autofagica dipendente da mTOR è necessaria per la Furthermore, mTOR-dependent autophagic action is required for ATO-induced NETosi
indotta da ATO nelle cellule APLs in APL cells [
129 ].
DaOf not
are che la rapamicina, l'ine, rapamycin, the inhibitor
e della via mTOR, si si of the mTOR pathway, synergiz
za con l'ATO nell'es with ATO in the eradica
zione delle cellule che iniziano la leucemiation of leukemia-initiating cells (LIC)
attraverso l'attivazione dellathrough the activation of NETosis
sia nelle cellulein both APL c
he in un modello APL in vivoells and an in vivo APL model [129
] .
Le tTra
slocazioni del gene della leucemia dinslocations of the mixed linea
mistge leukemia (
MLL ) 11q23
sogen
o state osservate in circa l'80% delle AML e are observed in approximately 80% of pediatric
he. In questi, AMLs. ilIn these, the MLL gene
MLLcan, può,by mediante traslocazione genomica, essere fuso congenomic translocation, be fused with >60 di
versifferent fusion partner
di fusiones [
130 ].
Il tTr
attamento con l'inibitore dell'eatment with the RAS oncogene
RASinhibitor, tipifamib,
porta all'inibizione dell'AML con la traslocazione t(6;11) inducendo sia l'aleads to inhibition of AML by the t(6;11) translocation inducing both apoptosi
che l'autofagias and autophagy [
131 ].
UAn
altro studio ha dimostrato che l'other study demonstrated that ATG5 part
ecipa allo sviluppo della leucemia guidata daicipates in the development of MLL-AF9- ,driven ma non nei topi chemioterapici sensibili all'AML che esprimonoleukemia , but not in AML-sensitive chemotherapy mice expressing MLL-AF9 [
132 ].
La leAcu
cemia mieloide acuta conte myeloid leukemia with core binding factor (
CBF-AML )
èis c
aratterizzata dalla haracterized by the presen
za dice of t(8;21) (q22; q22), o
r inv(16) (p13q22)/t(16;16),
che porta alla formazione dileading to the formation of RUNX1/RUNX1T1 (AML1/ETO) eand CBFbeta-MYH11 , r
iespe
ttivamentectively [
133 ].
L'attivazioneULK1-mediated activation of autophagy can control and delay AML1-ETO9a- d
ell'autofagri
a mediata da ULK1 può controllare e ritardare la leucven leukemogenesi
guidata das in an AML CASPAML1-ETO9aSE-3 ikn
unockout mouse model
lo murino [ knockout AML CASPASE-3 [ 134 ], sugge
rendo chesting that CASPASE-3
è uis an important
e regolatore dell'autofagia nell'AML. I risultati di questi regulator of autophagy in AML. The results of these studi
evidenziano i diversi ruoli dell'autofagia nell'inizio, nella es highlight the different roles of autophagy in the initiation, progression
e e nelle risposte chemioterapiche nelle cellule AML, a seconda del diverso tipo di and chemotherapy responses in AML cells, depending on the different type of aberrant oncoprotein
a aberrante.
4.2. MGene mutazioni genetiche itions in AML e autofagiaand autophagy
TraAmong le alterazionithe most common genetiche più comuni nell'AML, la mutazione del gene della tirosina chinasi alterations in AML, mutation of the tyrosine kinase 3 ( FLT3 ) sigene verifica in circa il 30% dei casi di AMLoccurs in approximately 30% of AML cases.
LThe
aberrazioni più most frequent
i che interessano il gen aberrations affecting the
FLT3 gene , associate
a unad with a poor prognosi
sfavorevole nella LMA, sono la duplicaziones in AML, are internal tandem
internaduplication (
FLT3 -ITD)
in
el dominio the juxtamembran
a e le mutazioni puntiformi, che coinvolgono il dominio tirosin-chinasico die domain and point mutations, involving the tyrosine kinase domain of FLT3 (
FLT3 -TKD) [
135 ]
.
L'esExpressione di of FLT3 -ITD
increa
umenta l'autofagia basale nelle cellule AML attraverso un meccanismo che coinvolge il fattore di trascrizioneses basal autophagy in AML cells through a mechanism involving the transcription factor ATF4 (a
ttivazione del fattore di trascrizionectivating transcription factor 4) [
136 ].
InFurthermo
ltre, l'inibizione dell'autofagia nellere, the inhibition of autophagy in cellule FLT3 cells-TKD,
which
e sono are resist
enti all'inant to the FLT3 inhibitor
e FLT3 quizartinib (AC220),
inibisce anche la also inhibits prolifera
zione siation both in vitro
cheand in vivo [
136 ].
PiùMore recent
emente, la mutazionely, the acquired D835Y
acquisita ha indottomutation induced resist
enza all'inance to the FLT3 inhibitor
e FLT3 sorafenib
e ha attivato l'autofagia nelle lineeand activated autophagy in cellulari FLT3 -ITD-positive
cell lines.
Inibendo l'autofagia, gli autori sono stati in grado di superare la resistenza al By inhibiting autophagy, the authors were able to overcome sorafenib
nella LMAresistance in FLT3 -ITD-positiv
ae AML,
migliorandone l'improving its efficac
iay [
137 ]. Recent
ementely,
uno studio ha dimostrato che l'inibizione dell'autofagia riduce il potenziale di ripopolamento delle LSC FLT3 -ITD AML assa study demonstrated that inhibition of autophagy reduces the repopulation po
ciate
all'accumulontial of mitoc
hondrial
e storage-associated [ FLT3 -ITD AML LSCs [ 138 ].
Inoltre, gli autori hanno dimostrato che l'inibizione dell'autofagia migliora l'attività di p53 e aumenta l'inibizione mediata da TKI dei Furthermore, the authors demonstrated that autophagy inhibition enhances p53 activity and enhances TKI-mediated inhibition of AML progenitor
i dell'AML s [
138 ].
L'aAuto
fagia non solophagy not only contribu
isce allates to the downstream prolifera
zione a valle del recettortion of the FLT3-ITD
, ma può anche essere coinvolta nella receptor but may also be involved in the degrada
zione del recettore mutato. Infatti, in uno studio, è stata osservata la tion of the mutated receptor. Indeed, in one study, the frequent
e attivazione del recettore tirosina chinasi RET activation of the RET receptor tyrosine kinase was observed in di
versi sottotipi di LMAfferent subtypes of AML [
139 ]. RET media
tes la sothe suppression
e dell'autofagia in modo di of autophagy in an mTORC1-dependent
e da mTORC1, portando alla manner, leading to the stabili
zzazione del recettore FLT3zation of the mutant
e. L'inibizione g FLT3 receptor. Genetic
a o f or pharmacologica
di RET ha ridotto la crescita delle cellule AML dil inhibition of RET reduced the growth of FLT3-dependent
i da FLT3, con la sovraregolazione dell'autofagia e la AML cells, with upregulation of autophagy and deple
zionetion diof FLT3 [
139].
QuThes
ti risultatie results sugge
riscono che il ripristino dell'autofagia nell'AML dist that restoration of autophagy in FLT3-dependent
e da FLT3 può comportare la AML may result in degrada
zione dell'FLT3 mutato e quindi può raption of mutant FLT3 and thus may represent
are un interessante approccio t an attractive therapeutic
o. È stato anche dimostrato che l'inibizione della proteina approach. Inhibition of the FLT3-ITD p
orta ad un aumento della sintesi dirotein has also been shown to lead to increased ceramide
e media la mitofagiasynthesis and mediate ceramide-d
iependent
e, portando alla morte delle cellule AML mitophagy, leading to AML cell death [140
, 141
] .
Le mMutazioni ditions in KIT sonoare associate
ad un'aumentatad with increased leukemia cell prolifera
zione di cellule leucemiche e ad un aumentato rischio di recidiva di AML tion and an increased risk of AML recurrence [
142 ,
143 ].
UnoA studio recent
e ha riportato che la mutazion study reported that the
KIT D816V
mutation
elle cellule AML aumenta l'autofagia basale in AML cells increases basal autophagy, stim
olando laulating AML cell prolifera
zione e la sopravvivenza delle cellule AML tramite la segnalazione STAT3 tion and survival via STAT3 signaling [
144 ].
UnaA di
versa mutazione puntiforme ifferent point mutation in c-
KIT (N822K T > A)
attiva co
nstitutiv
amente questo recettore, rendendo le cellule AML altamenteely activates this receptor, making AML cells highly sensi
bili altive to sunitinib (
un inibitore della tirosina chinasi), con conseguente morte cellulare AML attraverso l'attivazione sia dell'a tyrosine kinase inhibitor), resulting in AML cell death through the activation of both apoptosi
che deis and autophagic process
i autofagici es [
145 ]
.
Le mMuta
zionitions in
NPM1 (nucleophosmin 1)
sono le a
lterazioni genetiche piùre the most frequent
i nella LMA genetic alterations in adult
a AML, respons
abili dellaible for the aberrant locali
zzazione aberrante dellazation of the NPM1 protein
a NPM1 nel ci in the cytoplasm
a [
146 ].
L'auThe increased autophagic activity found in NPM1 -m
entata attività aut
ofagica riscontrata nelle cellule AML mutate con NPM1ated AML cells is è coinvol
ta nella sopravvivenza delle cellule leucemicheved in leukemic cell survival [
147 ].
Il mutantThe
NPM1 pmu
ò anchetant can also intera
gire con la proteina soct with the tumor suppressor
e tumorale protein PML (
proteinleukemia pro-m
ielocitica della leucemia), portando allayelocytic protein), leading to the delocali
zzazione e allazation and stabili
zzazione della PML che, a sua volta, può attivare l'autofagia tramite la segnalazione AKT zation of PML which, in turn, can activate autophagy via AKT signaling [147
] . In
uan
altro studio, è stato dimostrato che nei pazienti affetti da LMA portatori diother study, it was shown that in AML patients carrying the mutant
e NPM1 ,
l'enzima glicoliticothe glycolytic enzyme PKM2 (p
iruvato chinasi M2) ha indotto l'autofagia attraverso la fosforilazione della proteina autofagicayruvate kinase M2) induced autophagy through phosphorylation of the autophagic protein Beclin 1, contribu
endo alla sopravvivenza cellulareting to cell survival [
148 ].
InfFin
e, laally, the NPM1 mutant protein
a mutante NPM1 può anche can also intera
gire con la proteina autofagicact with the autophagic protein ULK1, stim
olando l'ulating TRAF6-dependent ubiquitina
zione dipendente da TRAF6 di ULK1 tramitetion of ULK1 via miR-146,
mantenendo così la thereby maintaining ULK1 stabilit
à e la funzy and functionalit
à di ULK1 e promuovendo la sopravvivenza delle cellule autofagichey and promoting autophagic cell survival [149
] .
InFurthermo
ltre, è stato osservato che l'esre, the expression
e di of RASGRP3,
una protein
a associat
a allaed with tumor progression
e del tumore, è sovraregolata nei pazienti, was observed to be upregulated in AML patients with conNPM1 LMA con muta
zionetion NPM1 compar
ispe
tto ai pazienti con LMA senzad to AML patients without mutant NPM1 mutant . The
. Gli aut
ori hanno dimostrato che hors demonstrated thatNPM1 -mut bloc
ca laks the degrada
zione della proteination of RASGRP3
attraverso il legame con la proteina MID1 dell'protein through binding to the E3 ubiquitin
a ligas
i E3, portando alla sovraese MID1 protein, leading to overexpression
e di of RASGRP3,
oltre a promuovere l'attivazione a valle di as well as promoting downstream activation of EGFR-STAT3,
che a sua volta promuove la which in turn promotes prolifera
zione e l'autofagia nelle cellule AML tion and autophagy in AML cells [150
] .
Altera
ztion
i del gene oncosos of the tumor suppressor
gene
TP53 siare riscontrano in circa ilfound in approximately 5-15%
dei casi di AML e, of AML cases and frequent
emente, nei pazienti più anziani ly in older patients [
151 ,
152 ].
ÈIt has been proposed that the role of autophagy in the development of AML may be determined by TP53 sta
to propost
o che il ruolo dell'autofagia nello sviluppo dell'AML possa essere determinato dallo stato di TP53 . Pus . For wild-type
r l'AML
TP53 wild-type ,
i ricercatori hanno dimostrato che il blocco fresearchers demonstrated that pharmacologic
o dell'autofagia ottiene benefici tal blockade of autophagy achieves therapeutic
i, mentre le AML che ospitano benefits, whereas AMLs harboring TP53 muta
ztion
is fail TP53 nto
n r
ispondono all'inibizione dell'autofagia da parte dell'idrossiclorochinaespond to autophagy inhibition by hydroxychloroquine (HCQ) [
153 ,
154 ].
L'uso di inThe use of autophagic inhibitor
i autofagici può essere uns may be a poten
ziale strategia tetial therapeutic
a da utilizzare, in strategy to use, partic
olare per il trattamento diularly for the treatment of wild-type TP53 AML
di tipo selvaggio. Per l'AML con. For AML with TP53 muta
ztion
is TP53 ,
le vie auto
fagiche possono essere un'opzione tphagic pathways may be a therapeutic
a da utilizzare per l' option to be used for the elimina
ziontion of the
delTP53 T mutant
e T TP53 .
UAn
altro studio ha dimostrato che la stimolazione dellaother study demonstrated that stimulation of macroauto
fagia da parte di phagy by 17-AAG,
un inan HSP90 inhibitor
e HSP90, provoca la, results in the degrada
zione dition of TP53 R248Q
nelle cellule AML e migliora anche la trascrizione dei geni in AML cells and also enhances the transcription of autophagy-associat
i all'autofagiaed genes [155
] .
InolFurt
re, le provehermore, accumulat
eing evidence indica
no chetes that TP53 a
ttivato da unctivated by a variet
à di stress y of cellular
i può innescare l'autofagia attraverso la stresses can trigger autophagy through the transa
ttivazione di geni ctivation of pro-auto
fagici, tra cuiphagic genes, including DRAM1DRAM1 ( DNA (dam
odulatore autofagico regolato dal danno al DNA age regulated autophagy modulator 1),
SESN1 (sestrin 1)
, e (and ( sestrin
SESN2 ) [
155 ,
156 ,
157 ,
158 ].
UnoA studio recent
e ha evidenziato il ruolo dell'autofagia nelle cellule AML, nel study highlighted the role of autophagy in AML cells, in the conte
sto dell'xt of p53-mediated apoptosi
mediata da p53, che ès, which is associat
a ad una maggiore citotossicità al trattamento con ined with increased cytotoxicity to treatment with MDM2 inhibitor
i MDM2 es and Ara-C
quando il when miR-10a
è inibitois inhibited [159
] .
La stratThe
gia antileu
cemica basata sull'uso di inibitorikemic strategy based on the use of MDM2/X
nei tumori p53 inhibitors in wild-type p
er ripristinare la conformazione normale e attiva di53 tumors to restore the normal and active conformation of p53, MDM2
e MDMX non è stata ampiamente testataand MDMX has not been extensively tested [160
] .
PTher
tanto, l'uso di unefore, the use of a combina
zione di trattamentition of treatments, inclu
si gli inding MDM2 inhibitor
i MDM2 cons with autophagic modulator
i autofagici, potrebbe essere una nuovas, could be a novel strateg
ia per migliorare il trattamento dell'AML p53 y to improve the treatment of wild-type
p53 AML .
I trPha
ttamenti farmacologic
i cheal treatments that modula
no l'autofagia nei pazienti con LMA portatorite autophagy in AML patients carrying dip53 muta
ztion
is p53 part
eicipa
no allate in the degrada
zione delle proteine p53 aberranti. La mutazione puntiformetion of aberrant p53 proteins. Point mutation of di TP53 nelat residuothe amino
acidico acid residue R428 (R248Q),
con attività di guadagno di funzione, dà origine ad attività maligna nelle cellule tumorali del polmone with gain-of-function activity, gives rise to malignant activity in lung cancer cells [
161 ]
e uan
a perdita della funzione di sod a loss of tumor suppress
ione tumorale nell'or function in AML [
162 ].
È iIntere
ssante notare che il trattamento con l'instingly, treatment with the Hsp90 inhibitor
e Hsp90 (17-AAG)
determina l'attivazione dell'autofagia results in the activation of chaperone-mediat
a da chaperone, cheed autophagy, which induce
las the degrada
zione della proteination of the aberran
tet protein p53R248Q
nelle cellule AML. In particolare, inin AML cells. Notably, under condi
zioni di stress tions of metabolic
o stress, 17-AAG induc
e l'es the intera
zione traction between p53R248Q
e la proteinaand the chaperone
protein Hsc70,
innescando l'autofagia triggering chaperone-mediat
a da chaperone pered autophagy to degrad
are p53R248Q [
155 ].
QuThes
ti dati aprono nuovee data open new opportunit
à peries for future studi
futuri che potrebbero chiarire il coinvolgimento funzionale di diversi tipi di autofagia e la loroes that could elucidate the functional involvement of different types of autophagy and their conne
ssione con i meccanismi molecolari per migliorare le tction with the molecular mechanisms for improving anticancer therapie
antitumorali contro l'AML che ospita le diverse s against AML harboring the different TP53 variant
is TP53 .
I rRecent
i progressi nella advances in bioinformatic
a hanno consentito l's have enabled the identifica
zione di diverse mutazionition of several epigenetic
he che interessano l'AML, tra cui mutations affecting AML, including IDH1/2 , Tet met
ihylc
itosina diossigenasiytosine dioxygenase 2 (
TET2 ), DNA met
ihyltransferas
ie 3A (
DNMT3A )
eand ASXL1 ,
tuttall of which are associate
alla patd with the pathogenesi
dell's of AML [
163 ,
164 ,
165].
LeIDH protein
e IDH sono s are isocitrat
o deie dehydrogenas
ies, implicate
d in vari
processious biologic
i, come il al processes, such as energy metabolism
o energetico, la demetilazione dell'istone, la, histone demethylation, DNA modifica
zione del DNA e l'adattamento all'ipossia. Sono necessari ulteriorition, and adaptation to hypoxia. Further studi
per studiare terapies are needed to investigate innovative
basate sull'autofagia mirata itherapies based on targeted autophagy in combina
zione con l'ipometilazione del DNA per trattare le AML che ospitano determinati tipi di alterazionition with DNA hypomethylation to treat AMLs harboring certain types of epigenetic
he alterations.
Mutazioni nel gene Mutations in the DNMT3A gene ,
un enzima coinvolto nella metilazione deian enzyme involved in CpG dinucleotid
i CpG, sonoe methylation, are present
i nel in 20-23%
dei pazienti adulti con AML of adult patients with de novo
AML [
165 ].
DiSever
sial studi
hanno dimostrato che il trattamento di pazienti affetti da LMA con agenti che inibiscono la es have shown that treatment of AML patients with DNA met
ihyltransferas
i, come l'e-inhibiting agents, such as azacitidin
ae (5-aza-2′-deo
ssicitidinaxycytidine), induce
l'attività autofagica nelle cellule di leucemia AMLs autophagic activity in AML leukemia cells [166
] .
UnoA stud
ioy in a DNMT3A R878H cond
oit
to su un modello murino ional knock-in
condizionale DNMT3A R878Hmouse model, u
sed t
ilizzato per prevedere glio predict the specific
i RNA lunghi non codificanti long noncoding RNAs (lncRNA
) regolatis) regulated by dalleDNMT3A muta
ztion
is in DNMT3A nell'AML,
ha pfir
imast identifi
catoed 23
lncRNAsdifferentially e
sxpress
i in modo differenziato , quindi i geni bersaglio a valle regolati da questied, then downstream target genes regulated by these lncRNA
s, inclu
soding ATP6V1A ,
un gene a critic
o correlato all'autofagia, la cui sovraesal autophagy-related gene, overexpression
e è of which is associat
a aed with poor prognosi
infausta nell's in AML [
167 ].
Tuttavia, ci sono ancora poche prove diHowever, there is still little evidence for a direct involvement of DNMT3A un coinvolg
imento diretto delle mutazioni del gene DNMT3A con attene mutations wi
vit
à autofagica nell'h autophagic activity in AML.
Sono necessaFuri ulteriorither studi per comprendere ies are needed to understand the functional significato funzionale dell'autofagiance of autophagy associata a diversed with different gene mutazioni genetiche nelle cellule AMLtions in AML cells.