AL'autophagy is a highly conservedfagia è un processo di degradazione cellular degradation process that regulates cellulare altamente conservato che regola il metabolism and ho cellulare e l'omeostasis underi in condizioni normal and pathophysiological conditions. Autophagy andi e fisiopatologiche. L'autofagia e il metabolism are linked in the ho sono collegati nel sistema ematopoietic system, playing a critical role in the self-renewal, survival ando, svolgendo un ruolo fondamentale nell'auto-rinnovamento, sopravvivenza e differentiation of hziazione delle cellule staminali e progenitrici ematopoietic stem and progenitor cells, and in cell death, he e nella morte cellulare, influenzando in particularly influencing the cell fate of the holare il destino cellulare del pool di cellule staminali ematopoietic stem cell pool. In leukhe. Nella leucemia, autophagy supports leukemia cell growthl'autofagia sostiene la crescita delle cellule leucemiche, contributes to leukemia stem cell survival andisce alla sopravvivenza delle cellule staminali leucemiche e alla resistance to chemotherapy. Acute myeloid leukemienza alla chemioterapia. La leucemia mieloide acuta (AML), a common type of acute leukemia with poor survival and prognosis un tipo comune di leucemia acuta con scarsa sopravvivenza e prognosi, è una malattia ematopoietica clonale che colpisce le cellule staminali e progenitrici ematopoietiche, che provoca il blocco della differenziazione mieloide e la soppressione delle funzioni ematopoietiche.
1. Autophfagy and Acute Myeloid Leukemia Stem Cellsia e cellule staminali AML
Dur
ante lo svi
ng leukemic development, stem cells (LSCs) can adapt theirluppo leucemico, le LSC possono adattare il loro metabolism
and autophagic mechanisms to provide the high energy ando e i meccanismi autofagici per fornire l'elevata energia e i nutrient
s required for LSCi necessari per la prolifera
tion and survival underzione e la sopravvivenza delle LSC in condi
tions ofzioni di carenza di nutrient
deficiency, starvation, hypoxia, or during chemotherapyi, fame, ipossia o durante i trattamenti chemioterapici [7 , 91 , 92 , 93 , 94 treatments] [1][2][3][4][5].
MLa mito
phagy, mitochfagia, la funzione mitocondrial
function and e e l'integrit
y can affect the viability, à possono influenzare la vitalità, la prolifera
tion andzione e il potenziale di differen
tiation potential andziazione e la longevit
y ofà delle normal
hi cellule staminali ematopoietic
stem cells (HSCshe (HSC)
[6][95] e ason
d are o important
in the survivali nella strateg
y of acute myeloid leukemia (AML) stem cells (LSCsia di sopravvivenza delle cellule staminali AML (LSC).
SIn p
ecifically, adenosine 5′-monophosphate (AMP)-activated protein kinase (AMPK), aarticolare, la proteina chinasi (AMPK) attivata dall'adenosina5′-monofosfato (AMP), un complesso protei
n complex critical inco fondamentale nel metabolismo mitoc
hondrial
metabolism and mitophagy, is cone e nella mitofagia, è costitutiv
ely activated in LSCs, increasingamente attivata nelle LSC, aumentando la clearance mitoc
hondrial
clearance to e per support
LSC growth and survival through its downstream targetare la crescita e la sopravvivenza delle LSC attraverso il suo obiettivo a valle FIS1,
thla fissione mitoc
hondrial
fission 1 e 1 componente protei
co di un comp
onent of alesso mitoc
hondrial
complex that promotese che promuove la fissione mitoc
hondrial
e [ 96 ]. La mitof
agi
ssiona mediata [7].da AMPK/FIS1
-m è nece
diated mitophagy is required for LSC self-renewal and survivalssaria per l'auto-rinnovamento e la sopravvivenza delle LSC [ [7]96].
OUna sov
erexraespression
ofe di FIS1
was also found in AML cells whileè stata trovata anche nelle cellule AML mentre la deple
tion ofzione di FIS1
impairs mitophagy, weakening the self-renewalcompromette la mitofagia, indebolendo la capacit
y of LSCs and resulting in the induction of myeloidà di auto-rinnovamento delle LSC e determinando l'induzione della differen
tiation by inactivation ofziazione mieloide mediante l'inattivazione di GSK3 (gl
ycogen synthase kinase 3), thus pointing to mitophagy as a regulatory mechanism for AMLicogeno sintasi chinasi 3), indicando così la mitofagia come meccanismo di regolazione per la progression
e [7]della LMA [ 96 ].
MorePiù recent
ly, the loss of emente, la perdita del sequestosom
ea 1 (
SQSTM1 ),
alsnoto
known as p62, a selectivanche come p62, un recettore auto
phagy receptorfagico selettivo crucial
for the development ande per lo sviluppo e la progression
of e dell'AML in vivo, induce
s the l'accumul
ation of damagedo di mitoc
hondria andondri danneggiati e superossido mitoc
hondrial
superoxide, thereby e, comprom
ising cell survival. leukemia cells. Thus, the lossettendo così la sopravvivenza delle cellule leucemiche. Quindi, la perdita di of SQSTM1i comp
airs leukemiromette la progression
in mouse models of AML, underscoring the role of mitophagy in LSC survivale della leucemia nei modelli murini di LMA, sottolineando il ruolo della mitofagia nella sopravvivenza delle [8]LSC.97 ]. Co
lmple
ctively, thesessivamente, questi studi
es demonstrate that increased autophagic activity of LSCs is required for malignant dimostrano che è necessaria una maggiore attività autofagica delle LSC per la progression
intoe maligna in AML.
HoweTuttav
er, in ia, contra
st to the activation of autophagy observed in AML, a loss of autophagy in healthy HSCs triggers the exriamente all'attivazione dell'autofagia osservata nell'AML, una perdita di autofagia nelle HSC sane innesca l'espansion
of a population ofe di una popolazione di cellule progenit
or cells in the bone marrow, resulting in severe and invasive myrici nel midollo osseo, dando origine a una mieloprolifera
tion, as in human AMLzione grave e invasiva, come nell'AML umana [53 ] [9].
ThiQues
to apparent
e parad
ox may be explained by the osso può essere spiegato dai ruoli distin
ct roles that autophagy may play during AMLti che l'autofagia può svolgere durante la progression
, which may be different at various stages of leuke dell'AML, che può essere diversa nelle varie fasi della leucemogenesi
s [ 98 , [10][11]99].
AL'auto
phagy in normal HSCs mayfagia nelle HSC normali può preven
t the onset of cancer, as a tumor suire l'insorgenza del cancro, come meccanismo soppressiv
e mechanism. Indeed, autophagy removes damagedo del tumore. Infatti, l'autofagia rimuove gli organell
es, such asi danneggiati, come i mitoc
hondria, andondri, e prote
cts hgge le cellule ematopoietic
cells from genomic ihe dall'instabilit
y and inflammation, thus à genomica e dall'infiammazione, preven
ting the onset of leukendo così l'insorgenza della leucemia.
Notably, increased DNA damage, high ROS levels, In particolare, l'aumento del danno al DNA, gli alti livelli di ROS, l'aneuploid
y, and ania e un accumulo aberrant
accumulation of e di p62/SQSTM1
have been sono stati correlat
ed with an impaired autophagy processi con un processo autofagico alterato, indica
ting a key role of autophagy inndo un ruolo chiave dell'autofagia nella preven
ting the initiation ofzione dell'inizio del tumor
e [100 [12]] .
CAl con
versely, in established cancer, autophagy may function as atrario, nel cancro accertato, l'autofagia può funzionare come un percorso favor
able pathway that promotesevole che promuove la sopravvivenza e la crescita del tumor
survival and growth,
Somee, aiutando le cellule tumorali a sfuggire allo studires have also ss metabolico e agli stimoli di morte.
Alcuni s
tudi h
own that the activation of the autophagic flux only plays a role in the initiation of AML, with a trananno anche dimostrato che l'attivazione del flusso autofagico gioca un ruolo solo nell'inizio dell'AML, con una trasforma
tion from HSC tozione da HSC a LSC,
and, therefore, after this stage, autophagy is not required for disease maintenancee, quindi, dopo questa fase, l'autofagia non è necessaria per il mantenimento della [13]malattia [101 ] .
AMLGli studi
es sull'AML MLL-AF9,
the most common l'altera
tion in childhood AML, point tzione più comune nell'AML infantile, indicano ATG5 o
r ATG7
as required for the onset of AML, but once the leukemiccome necessari per l'insorgenza dell'AML, ma una volta stabilita la condi
tion is established, autophagy is not required for functionzione leucemica, l'autofagia non è richiesta per la funzione LSC in vivo
[13][14][101 , 102 ] .
HoweTuttav
er, in a differentia, in un diverso modello di topo AML MLL-ENL
mouse model, , il knockout Atg5 o
r Atg7
knockout reduced the number of functional LSCs, increased theha ridotto il numero di LSC funzionali, aumentato l'attivazione mitocondriale e i livelli di ROS in queste cellule e prolungato la sopravvivenza dei topi leucemici [15][103] .]. In
quest
hiso conte
xt, during the sto, durante il process
of leuko di leucemogenesi
s, histone methylation can regulate central autophagic effectors and upstream autophagic regulators such as , la metilazione dell'istone può regolare gli effettori autofagici centrali e i regolatori autofagici a monte come ATG 5
ande ATG7
toper influenzare indire
ctly affect the level of autophagyttamente il livello di autofagia [ 104 [16]].
TogInsie
ther, theseme, questi studi
es sugge
st a highlyriscono un ruolo altamente comple
x and context-desso e dipendent
role for autophagy in leukemic trane dal contesto per l'autofagia nella trasforma
tion with respect to the maintenance properties of LSCs in zione leucemica rispetto alle proprietà di mantenimento delle LSC nell'AML.
TheIl du
al role of autophagy in AML, as a plice ruolo dell'autofagia nell'AML, come promot
er or suore o soppressor
of cancer in AML, is still a matter of debate. Studies have demonstrated that autophagy can act as a pro- ore del cancro nell'AML, è ancora oggetto di dibattito. Gli studi hanno dimostrato che l'autofagia può agire come meccanismo pro o anti-proliferativ
e mechanism depending on the lineage and molecularo a seconda del lignaggio e del contesto genot
ypic context of the disease, reflecting the degree of hipico molecolare della malattia, riflettendo il grado di eterogeneit
y of à dell'AML
[17][105 ] .
2. Reguolation of Autophagy Genes in Acute Myeloid Leukemia Cellszione dei geni dell'autofagia nelle cellule AML
Numerousi studies have demonstrated that increased autophagy in AML cells hanno dimostrato che l'aumento dell'autofagia nelle cellule AML confersisce protection from chemotherapy treatment and promotes AML cell survival.
Inzione dal trattamento chemioterapico e promuove la sopravvivenza delle creased ATG7-llule AML.
L'aume
dintat
ed autophagy has beena autofagia mediata da ATG7 è stata associat
ed with poora a scarsi risultati clinic
al outcomes and shorti e a una breve durat
ion ofa della remission
in AML patientse nei pazienti con LMA [ 106 [18]].
MorePiù recent
ly, somemente, alcune protein
s involved in leukemia cell survival and overexe coinvolte nella sopravvivenza delle cellule leucemiche e sovraespresse
d in AML have been nell'AML sono state correlate
d to ATG overex alla sovraespression
, underlying the interplay between autophagy and protein overexe di ATG, alla base dell'interazione tra autofagia e sovraespression
that promotes leukemia cell survivale proteica che promuove la sopravvivenza delle cellule leucemiche [8] [19].]. Hu et al.
hanno d
emonstrated that imostrato che un'elevat
ed exa espression
ofe di SIRT1 (Sirtuin 1),
a key player in un attore chiave nella biogenesi mitoc
hondrial
biogenesis and autophagy-related protein, ise e nella proteina correlata all'autofagia, è associat
ed with a a un'elevat
ed exa espression
ofe di CXCR4,
a negativeun marker prognostic
marker in AML, and othero negativo nell'AML e ad altre protein
s e correlate
d to autophagy such as all'autofagia come ATG5
ande LC3 in
primary human AML samplescampioni umani primari di AML, indica
ting ando un ruolo poten
tial role of thziale della via di segnalazione SDF-1α-CXCR4
signaling pathway in the induction of autophagy in AML cells, which further promotes their survival undernell'induzione dell'autofagia nelle cellule AML, che promuove ulteriormente la loro sopravvivenza sotto stress
[20][107 ] .
ThIl canale ionico del recettore trans
ientitorio della melastatin
receptor a 2 (TRPM2)
ion channel, involved in the maintenance of cell survival after ox, coinvolto nel mantenimento della sopravvivenza cellulare dopo il danno ossidativ
e damage, is highly expressed in AMLo, è altamente espresso nell'AML [ 108 [21]].
By performEseguendo l'esaurimento ding TRPM2 depletion , Chen SJ et al.
[ [21]108 ] hanno d
eimo
nstrated that the levels of strato che i livelli di proteine ULK1, Atg7
ande Atg5
proteins are decreased insono diminuiti nelle cellule impoverite di TRPM2- , dep
leted cells , leading to inhibition of autophagy. Iortando all'inibizione dell'autofagia. È important
ly,e sottolineare che l'esaurimento di TRPM2 dne
pletion in ll'AML in
hibits leukemiibisce la prolifera
tion and increases thezione della leucemia e aumenta la sensibilità alla doxorubicin
sensitivity of AML cellsa delle cellule AML [ 108 [21]].
FStudi fun
ctional studies in normal CD34+CB cellszionali su normali cellule CD34 + CB hanno indicat
ed that inhibition of VMP1 exo che l'inibizione dell'espression
reduced autophagic flux, with decreased hematopoietic stem ande di VMP1 ha ridotto il flusso autofagico, con diminuzione dell'espansione delle cellule staminali e progenit
or cellrici ematopoietiche (HSPC)
expansion, delayed d, differen
tiation, increased ziazione ritardata, aumento dell'apoptosi
s, and impaired cellular function and engraftment e compromissione della funzione cellulare e dell'attecchimento in vivo.
Similar results were observed in leukemic cell lines andRisultati simili sono stati osservati nelle linee cellulari leucemiche e nelle cellule AML CD34+ primar
y CD34+ AML cells. Furthermore, ie. Inoltre, l'analisi ultrastru
ctural analysistturale ha indicat
ed that leukemic cells overexpressingo che le cellule leucemiche che sovraesprimono VMP1 ha
ve a reduced number of mitochondrial structures and the number of lysosomalnno un numero ridotto di strutture mitocondriali e il numero di strutture di degrada
tion structures is increased. Overexzione lisosomiale è aumentato. La sovraespression
ofe di VMP1 (vacuole membrane protein-1)
increased autophagic flux and improved mitochha aumentato il flusso autofagico e migliorato la qualità mitocondriale, che ha coinciso con un aumento della soglia per la perdita indotta da venetoclax della permeabilizzazione della membrana esterna mitocondrial
qualitye (MOMP) e dell'apoptosi nelle cellule leucemiche.109 [22]].
HDelezioni eteroz
ygous deletions,igoti, mutazioni missens
e mutations, or changes in copy number of key autophagy genes have been found with a high o o cambiamenti nel numero di copie dei geni chiave dell'autofagia sono stati trovati con un'alta frequen
cy in AML patients, za nei pazienti con LMA, in partic
ularly in AML patients with complex karyotypesolare nei pazienti con LMA con cariotipi complessi [5 , 103 ] [15][23]. In partic
uolar
, a heterozygous che, una perdita cromosom
al loss ofica eterozigote di 5q, 16q
, or 17p o 17p è correlat
es with a alle region
s encoding the autophagici che codificano i geni autofagici genes ATG10 ande ATG12 ,
GABARAPL2 ,e and MAP1LC3B o
r GABARAP , r
eispe
ctivelyttivamente [ 103 [15]],
ande m
any other autophagic genes have a low level of exolti altri geni autofagici hanno un basso livello di espression
in the Human AML blasts, a reduced autophagic flux, and high levels ofe nell'AML umano blasti, un flusso autofagico ridotto e alti livelli di ROS
[15][ 103 ].
FurInolt
hermore, one studyre, uno studio ha sugge
sted that key autophagy grito che i geni chiave dell'autofagia come
nes such as ULK1 ,
ATG3 ,
ATG4D ande ATG5 wer
eano signific
antly downregulated inativamente sottoregolati nelle cellule AML primar
y AML cells compared to ie rispetto ai normal
i granuloc
ytesiti [ 110 [24]].
3. ABiomarcatori autophagic Bfagiciomarkers
SigRecentemente son
ificanto stati compiuti progress
has recently been made to i significativi per identif
yicare specific
autophagy-related genes for the prediction of i geni correlati all'autofagia per la previsione degli esiti clinic
al outcomes in AML.i nell'AML. Insieme ai geni Along with the previously described ATG gprecedentemen
es , severalte descritti, diversi microRNA
s implicat
ed in leuki nella leucemogenesi
s and chem e nella chemioresist
ance have also been involved in autophagy activation and can be used asenza sono stati coinvolti anche nell'attivazione dell'autofagia e possono essere utilizzati come biomar
kerscatori [ 111 [25]].
NIn partico
tably, miR-17-5p overexlare, la sovraespression
in leuke di miR-17-5p nella leucemia prom
otes AMLuove la prolifera
tion by inhibiting autophagy through BECN1zione di AML inibendo l'autofagia attraverso il targeting
[26][27][28]BECN1 [ 112 , 113 , 114 ]. Gansan et al. d
eimo
nstrated thatstrato che le cellule stromal
cells downregulate i sottoregolano i livelli di miR-23a-5p
levels in leukemia cells, leading to upregulation ofnelle cellule leucemiche, portando alla sovraregolazione dell'autofagia prote
ctive autophagy in thesttiva in queste cell
s, thereby increasing theirule, aumentando così la loro resist
ance to chemotherapyenza alla tossicità della chemioterapia.115 ]. È st
oxicityato dimostrato che [29].la Osov
erexraespression
ofe di MiR-143
has been shown to enhance the sensitivity of AML cells to the cytotoxicity of cymigliora la sensibilità delle cellule AML alla citotossicità del trattamento con citarabin
ea (Ara-C)
treatment by inhibiting autophagy throughinibendo l'autofagia attraverso il targeting ATG7
ande ATG2B
[ [30]116 ].
AUn
overexa sovraespression
ofe di miR-15a-5p
is involved in the chemè coinvolta nella chemioresist
ance of AML patients, through the autophagy-relatedenza dei pazienti con LMA, attraverso i geni correlati all'autofagia genes ATG9A ,
ATG14 ,
GABARAPL1 ande SMPD1whi ch
target AML cellse prendono di mira le cellule AML [ 117 [31]].
RI recent
advances ini progressi nella bioinformatic
s have yielded an autophagy-related signature that may help predict overall survivala hanno prodotto una firma correlata all'autofagia che può aiutare a prevedere la sopravvivenza globale (OS)
and/ore/o gli esiti clinic
al outcomes of patients with AML. Severali dei pazienti con LMA. Diversi studi
es have shown that the hanno dimostrato che la progression
of AML depends on the gene signaturee dell'AML dipende dalla firma genica associat
ed with autophagya all'autofagia [ 118 [32]].
AUn recent
e studio di bioinformatic
s study constructed a model containing 10 autophagy-related genes to predict survival of AML patients, demonstrating that groups at high AML risk have higher exa ha costruito un modello contenente 10 geni correlati all'autofagia per prevedere la sopravvivenza dei pazienti affetti da AML, dimostrando che i gruppi ad alto rischio di AML hanno una maggiore espression
of immunee di geni del checkpoint
genes and a higher immunitario e una percent
age ofuale più alta di cellule CD4 T
and NK cellse NK [119] [33].].
FurInolt
hermore, this research was able tore, questo studio è stato in grado di predi
ct OS in AML through the signature ofre l'OS nella LMA attraverso la firma di 10 gen
es, pointing to this model as an effectivi, indicando questo modello come un efficace predittore prognostic
predictor for AML patients, useful to guide patiento per i pazienti con LMA, utile per guidare la stratifica
tion fozione dei pazienti per immunot
herapie
s and medications e farmaci [119 ] [33].
The L
ASSO Coxo studio di bioinformatic
sa regression
study whiche LASSO Cox che ha identifi
ed a critical risk signature focato una firma di rischio critico per AML, co
nsisting of the autophagicstituito dai geni autofagici genes BAG3 ,
CALCOCO2 ,
CAMKK2 ,
CANX ,
DAPK1 ,
P4HB ,
TSC2 ande ULK1 ,
haveva
d ex un eccellent
predictive power for AML e potere predittivo per la prognosi
s AML [ [34]120].
NIn partico
tably,lare, le citochine immunos
uoppressive
cytokines were found to bsono risultate significa
ntly increased in the tumortivamente aumentate nel micro
environment of patients with a high risk of ambiente tumorale di pazienti con un alto rischio di AML, pre
dicted on the basis of the autophagy-related signature of these patientsvisto sulla base della firma correlata all'autofagia di questi pazienti [120 ] [34].
HoweTuttav
er, thia, il valore prognostic
value of the ATG signature in theo della firma ATG in ambito clinic
al setting is still debated. Therefore, the roles of the ATG signature and autophagy in the patho è ancora dibattuto. Pertanto, i ruoli della firma ATG e dell'autofagia nella patogenesi
s of AML should be further investigated dell'AML dovrebbero essere ulteriormente studiati.
FurInolt
hermore, an interesting studyre, uno studio interessante ha indicat
ed that an autophagy-relatedo che una firma di lncRNA
signature containing sixcorrelata all'autofagia contenente sei lncRNA
s (
HYMAI ,
MIR155HG ,
MGC12916 ,
DIRC3 ,
C1orf220 ande HCP5 )
maypuò have aavere un important
e valore prognostic
valueo [ 121 [35]].
AUno studio recent
study pointed to four autophagy-ae ha indicato quattro lncRNA associat
ed lncRNAsi all'autofagia (
MIR133A1HG ,
AL359715.1 ,
MIRLET7BHG , ande AL356752.1 )
come firma
s signatures to da utilizzare poten
tially be used as a zialmente come biomar
ker to predict survival of AML patientscatore per predire la sopravvivenza dei pazienti con LMA [ 122 [36]].
Compllectively, these dataessivamente, questi dati indicate that the role of autophagy inno che il ruolo dell'autofagia nello sviluppo del tumor development clearly depends on the type of AML and stage of tumor development. Furthermore, autophagy may provide cancer cells with a survival strategye dipende chiaramente dal tipo di LMA e dallo stadio di sviluppo del tumore. Inoltre, l'autofagia può fornire alle cellule tumorali una strategia di sopravvivenza, suggesting a therendo un uso terapeutic use for autophagy inhibition. On the other hand, autophagy can induco per l'inibizione dell'autofagia. D'altra parte, l'autofagia può indurre la morte cell death, pointing to autophagy activation as a novelulare, indicando l'attivazione dell'autofagia come una nuova strategy in cancer therapy. Therefore, it is ia nella terapia del cancro. Pertanto, è necessary tio determine the role played by autophagy in theare il ruolo svolto dall'autofagia nei sottotipi molecular subtypes of olari di AML, or the degree of tumor development, to il grado di sviluppo del tumore, per verify whether itsicare se la sua modulation could lead to benefits for the treated patientzione potrebbe portare a benefici per il paziente trattato.
4. Autophfagy and Genetic Alterations in Acute Myeloid ia e alterazioni genetiche nell'AMLeukemia
ThIl fe
AML phenotype results from multiplenotipo AML deriva da molteplici alterazioni molec
ularolari, genetic
, anhe ed epigenetic
alterations that affect the he che influenzano la differen
tiation, ziazione, la prolifera
tion, and zione e l'apoptosi
s of myeloid dei progenitor
s. The World Health i mieloidi. L'Organi
zation haszzazione Mondiale della Sanità ha classifi
ed AMLs based on the cato le AML in base alla presen
ce ofza di partic
ular geneticolari altera
tions, oftenzioni genetiche, spesso originat
ing from chromosomal translocations or other genome ree da traslocazioni cromosomiche o altri riarrang
ements such asiamenti del genoma come t(8;21), t(15;17), inv(16), inv
(3) , t(6;9), t(9;11) o
r t(11;19), o
r mutations in receptor kinases, key signaling mediators mutazioni nelle chinasi del recettore, in mediatori di segnalazione chiave, proto-oncogen
es, ori o enzimi epigenetic
enzymes, for examplei, ad esempio, muta
tionszioni in FLT3 (FMS
- like t
yirosin
e kinasea chinasi 3),
TP53 ,
c-KIT o
r IDH1/2 ,
NPM1 (nucleo
phosmin 1) and CCAAT enhancer binding proteinfosmina 1) e proteina legante potenziatore CCAAT (
CEBPA )
[37][38][39][1 , 2 , 123 ].
ThQues
te muta
tions inzioni nelle AML ha
ve anno un impa
ct on the choice of the most suitable therapytto sulla scelta della terapia più adatta.
The L'associa
tion between autophagy and recurrentzione tra autofagia e alterazioni genetic
alterations has been he ricorrenti è stata descri
bed in several AMLtta in diversi studi
es, but needs further investigation sull'AML, ma necessita di ulteriori indagini [ 124 , [40][41]125 ].
HereQui, r
esearchers summarize and updateiassumiamo e aggiorniamo i recent
advances that have highlighted the link between autophagy andi progressi che hanno evidenziato il legame tra autofagia e geni di fusion
genes and recurrent e e mutazioni oncogenic
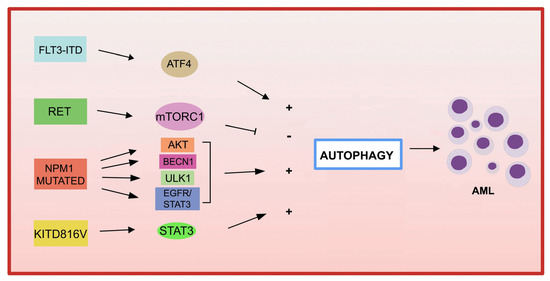
mutations in AML and the involvement of autophagy in chemotherapy treatmenthe ricorrenti nell'AML e il coinvolgimento dell'autofagia nel trattamento chemioterapico ( (Figurea 1 ).
Figurea 1. Gene mMutations in AML and autophagy. Several recurrentzioni genetiche nell'AML e nell'autofagia. Diverse anomalie genetic abnormalities in AML are involved in thehe ricorrenti nell'AML sono coinvolte nella deregulation of autophagy, leading to leukemiolazione dell'autofagia, portando alla progressione della leucemia.
4.1. Fusion Genes in Acute Myeloid Leukemia and Autophagy
4.1. Geni di fusione in AML e autofagia
MLa maggio
st cases of APL are caused by a de novo translocation tr parte dei casi di APL sono causati da una traslocazione de novo t (15;17)
(q22;q21), resulting in the (q22; q21), che risulta nella fusion
of the del gene
RARA gecon
e with th il gene
PML gene[ 126 , 127 [42][43]].
APL
cells that have lower exe cellule APL che hanno un'espression
of autophagy-related genes thane inferiore di geni correlati all'autofagia rispetto alle cellule normal
cells have reduced autophagic activity.i hanno un'attività autofagica ridotta. Utilizzando agenti differenzianti, come l'acido all Using differentiating agents, such as all- trans- retinoid
e acid (ATRA)
ande il triossido di arsenic
trioxideo (ATO)
currently used in attualmente utilizzati in ambito clinic
al settings, the exo, il livello di espression
level of autophagy-related genes increases, thus restoring autophagy in APL cellse dei geni correlati all'autofagia aumenta, ripristinando così l'autofagia nelle cellule APL [ [44]128].
BoEnt
hrambi gli agent
s can activate i possono attivare l'ETosis,
a type of cell deathun tipo di morte cellulare mediat
ed by the release of neutrophila dal rilascio di trappole extracellular
trapsi di neutrofili (ET).
Furthermore, mTOR-dependent autophagic action is required for ATO-inducedInoltre, l'azione autofagica dipendente da mTOR è necessaria per la NETosi
s in APL cells indotta da ATO nelle cellule APL [ 129 [45]].
OfDa not
e,are che la rapam
ycin, the inhicina, l'inibitor
of the mTOR pathway, synergizes with ATO in the e della via mTOR, si sinergizza con l'ATO nell'eradica
tion of leukemia-initiating cellszione delle cellule che iniziano la leucemia (LIC)
through the activation ofattraverso l'attivazione della NETosis
in bothsia nelle cellule APL c
ells and anhe in un modello APL in vivo
APL[129 model] [45].
TLe tra
nslocations of the mixedslocazioni del gene della leucemia di linea
ge leukemi mista (
MLL ) 11q23
gene are observed in approximately 80% ofsono state osservate in circa l'80% delle AML pediatric
AMLs.he. In questi, In these, the MLLil gene
canMLL può,
by gme
nomic translocation, be fused withdiante traslocazione genomica, essere fuso con >60 di
fferent fusionversi partner
s di fusione [46][ 130 ].
TIl tr
eatment with the RAS attamento con l'inibitore dell'oncogene
inhibitorRAS, tipifamib,
leads to inhibition of AML by the t(6;11) translocation inducing both porta all'inibizione dell'AML con la traslocazione t(6;11) inducendo sia l'apoptosi
s and autophagy che l'autofagia [ 131 [47]].
AUn
other study demonstrated that altro studio ha dimostrato che l'ATG5 part
icipates in the development ofecipa allo sviluppo della leucemia guidata da MLL-AF9- driven, leukemia , but not in AML-sensitive chemotherapy mice expressingma non nei topi chemioterapici sensibili all'AML che esprimono MLL-AF9
[ [47]132 ].
ALa leuc
ute myeloid leukemia withemia mieloide acuta con core binding factor (
CBF-AML )
is characterized by the è caratterizzata dalla presen
ce ofza di t(8;21) (q22; q22), o
r inv(16) (p13q22)/t(16;16),
leading to theche porta alla forma
tion ofzione di RUNX1/RUNX1T1 (AML1/ETO) ande CBFbeta-MYH11 , r
espeispettivamente [ 133 ]. L'attivazione dell'autofagia mediata da ULK1 può c
ont
ivelyrollare e ritardare la leucemogenesi guidata [48].da ULK1-mediated activation of autophagy can control and delay AML1-ETO9a- dri
ven leukemogenesis in ann un modello murino knockout AML
CASPASE-3 [ 134 knockout mouse model [49]], sugge
stiren
g thatdo che CASPASE-3
is aè un important
regulator of autophagy in AML. The results of thesee regolatore dell'autofagia nell'AML. I risultati di questi studi
es highlight the different roles of autophagy in the initiation, evidenziano i diversi ruoli dell'autofagia nell'inizio, nella progression
and chemotherapy responses in AML cells, depending on the different type of aberrante e nelle risposte chemioterapiche nelle cellule AML, a seconda del diverso tipo di oncoprotein
a aberrante.
4.2. Gene Mutations in Acute Myeloid Leukemia and Autophagy
4.2. Mutazioni genetiche in AML e autofagia
FLT3
AmongTra the most commonle alterazioni genetic alterations in AML, mutation of the tyrosine kinasehe più comuni nell'AML, la mutazione del gene della tirosina chinasi 3 ( FLT3 ) gsi vene occurs in approximately 30% of AML casesrifica in circa il 30% dei casi di AML.
ThLe
mostaberrazioni più frequent
aberrations affecting thi che interessano il gene
FLT3 gene , associate
d with a poor pr a una prognosi
s in AML, are internal sfavorevole nella LMA, sono la duplicazione tandem
duplicationinterna (
FLT3 -ITD)
nel domin
the io juxtamembran
e domain and point mutations, involving the tyrosine kinase domaina e le mutazioni puntiformi, che coinvolgono il dominio tirosin-chinasico di of FLT3 (
FLT3 -TKD)
[ [50]135 ] .
ExL'espression ofe di FLT3 -ITD
iaumen
creases basal autophagy in AML cells through a mechanism involving the transcription factor ta l'autofagia basale nelle cellule AML attraverso un meccanismo che coinvolge il fattore di trascrizione ATF4 (a
ctivating transcription factor ttivazione del fattore di trascrizione 4)
[51][ 136 ].
FurInolt
hermore, the inhibition of autophagy inre, l'inibizione dell'autofagia nelle cellule FLT3 cells -TKD,
which
aree sono resist
ant to thenti all'inibitore FLT3
inhibitor quizartinib (AC220),
also inhibits inibisce anche la prolifera
tion bothzione sia in vitro
andche in vivo
[51][ 136].
MorePiù recent
ly, the acquiredemente, la mutazione D835Y
mutation inducedacquisita ha indotto resist
ance to thenza all'inibitore FLT3
inhibitor sorafenib
and activated autophagy in e ha attivato l'autofagia nelle linee cellulari FLT3 -ITD-positiv
e cell line
s. By inhibiting autophagy, the authors were able to overcome. Inibendo l'autofagia, gli autori sono stati in grado di superare la resistenza al sorafenib
resistance innella LMA FLT3 -ITD-positiv
e AMLa,
improving its migliorandone l'efficac
yia [ 137 [52]]. Recent
lyemente,
a study demonstrated that inhibition of autophagy reduces the repopulationuno studio ha dimostrato che l'inibizione dell'autofagia riduce il potenziale di ripopolamento delle LSC FLT3 -ITD AML passo
tential ofciate all'accumulo mitoc
hondrial
storage-associatede [ 138 FLT3 -ITD AML LSCs [53]].
FurInolt
hermore, the authors demonstrated that autophagy inhibition enhances p53 activity and enhances TKI-mediated inhibition of AMLre, gli autori hanno dimostrato che l'inibizione dell'autofagia migliora l'attività di p53 e aumenta l'inibizione mediata da TKI dei progenitor
si dell'AML [ 138 [53]].
AL'auto
phagy not onlyfagia non solo contribu
tes to the downstreamisce alla prolifera
tion of thzione a valle del recettore FLT3-ITD
receptor but may also be involved in the, ma può anche essere coinvolta nella degrada
tion of thzione del recettore mutat
ed receptor. Indeed, in one study, the o. Infatti, in uno studio, è stata osservata la frequent
activation of the RET receptor tyrosine kinase was observed in different subtypes of AMLe attivazione del recettore tirosina chinasi RET in diversi sottotipi di LMA [ 139 [54]]. RET media
tes the sula soppression
of autophagy in an mTORC1-dee dell'autofagia in modo dipendent
manner, leading to thee da mTORC1, portando alla stabili
zation of the zzazione del recettore FLT3 mutant
FLT3 receptor. Ge. L'inibizione genetic
or pha o farmacologica
l inhibition of RET reduced the growth of FLT3-de di RET ha ridotto la crescita delle cellule AML dipendent
AML cells, with upregulation of autophagy andi da FLT3, con la sovraregolazione dell'autofagia e la deple
tionzione ofdi FLT3 [ [54]139].
ThQues
e resultsti risultati sugge
st that restoration of autophagy in FLT3-deriscono che il ripristino dell'autofagia nell'AML dipendent
AML may result in e da FLT3 può comportare la degrada
tion of mutant FLT3 and thus may rezione dell'FLT3 mutato e quindi può rappresent
an attractive thare un interessante approccio terapeutic
approach. Inhibition of theo. È stato anche dimostrato che l'inibizione della proteina FLT3-ITD p
rotein has also been shown to lead to increasedorta ad un aumento della sintesi di ceramide
synthesis and mediatee media la mitofagia ceramide-d
eipendent
mitophagy, leading to AML cell deathe, portando alla morte delle cellule AML [140 , 141 ] [55][56].
KIT
MLe mutations inzioni di KIT aresono associate
d with increased leukemia cell proliferation and an increased risk of AML ad un'aumentata proliferazione di cellule leucemiche e ad un aumentato rischio di recidiva di AML [ recurrence142 , 143 [57][58]].
AUno studio recent
study reported that the ha riportato che la mutazione
KIT D816V
mutation
in AML cells increases basal autophagyelle cellule AML aumenta l'autofagia basale, stim
ulating AML cellolando la prolifera
tion and survival via STAT3 signalingzione e la sopravvivenza delle cellule AML tramite la segnalazione STAT3 [ 144 [59]].
AUna di
fferent point mutationversa mutazione puntiforme in c-
KIT (N822K T > A)
attiva co
nstitutiv
ely activates this receptor, making AML cells highlyamente questo recettore, rendendo le cellule AML altamente sensi
tive tobili al sunitinib (
a tyrosine kinase inhibitor), resulting in AML cell death through the activation of both un inibitore della tirosina chinasi), con conseguente morte cellulare AML attraverso l'attivazione sia dell'apoptosi
s and autophagic processes che dei processi autofagici [ 145 ] [60].
NPM1
MLe muta
tionszioni in
NPM1 (nucleophosmin 1)
sono are the mostle alterazioni genetiche più frequent
genetic alterations ini nella LMA adult
AMLa, respons
ible for the aberrantabili della localiz
ation of the NPM1zazione aberrante della protein
in the cya NPM1 nel citoplasm
a [61][ 146 ].
The increased autophagic activity found in NPM1L'aumentata attività -maut
ated AML cellsofagica riscontrata nelle cellule AML mutate con NPM1 isè involved in leukemic cell survivalcoinvolta nella sopravvivenza delle cellule leucemiche [ 147 [62]].
ThIl mutante
NPM1 mpu
tant can also ò anche intera
ct with the tumor sugire con la proteina soppressor
proteine tumorale PML (
leukemiproteina pro-m
yelocytic protein), leading to theielocitica della leucemia), portando alla delocali
zation andzzazione e alla stabiliz
ation of PML which, in turn, can activate autophagy via AKT signalingzazione della PML che, a sua volta, può attivare l'autofagia tramite la segnalazione AKT [147 ] [62]. In
un a
nother study, it was shown that in AML patients carrying theltro studio, è stato dimostrato che nei pazienti affetti da LMA portatori di mutant
e NPM1 ,
the glycolytic enzyme l'enzima glicolitico PKM2 (p
yruvate kinase M2) induced autophagy through phosphorylation of the autophagic protein iruvato chinasi M2) ha indotto l'autofagia attraverso la fosforilazione della proteina autofagica Beclin 1, contribu
ting to cell survivalendo alla sopravvivenza cellulare [63][ 148 ].
FInfin
ally, the NPM1 e, la proteina mutant
protein can also e NPM1 può anche intera
ct with the autophagic protein gire con la proteina autofagica ULK1, stim
ulating TRAF6-dependent olando l'ubiquitina
tion ofzione dipendente da TRAF6 di ULK1
viatramite miR-146,
thereby maintaining ULK1mantenendo così la stabilit
y and functà e la funzionalit
y and promoting autophagic cell survivalà di ULK1 e promuovendo la sopravvivenza delle cellule [64]. Fau
rt
hermofagiche [149 ] . Ino
ltre,
the exè stato osservato che l'espression
ofe di RASGRP3,
una protein
a associat
ed with tumor a alla progression
, was observed to be upregulated in AML patients withe del tumore, è sovraregolata nei pazienti con LMA NPM1con muta
tzion
e comNPM1 risp
are
d to AML patientstto ai pazienti con LMA senza withoutNPM1 mutant
e . NPM1. TheGli aut
ori h
ors demonstratedanno dimostrato che thatNPM1 -mut blo
cks thecca la degrada
tion ofzione della proteina RASGRP3
protein through binding to the E3 attraverso il legame con la proteina MID1 dell'ubiquitin
a ligas
e MID1 protein, leading to overexi E3, portando alla sovraespression
ofe di RASGRP3,
as well as promoting downstream activation ofoltre a promuovere l'attivazione a valle di EGFR-STAT3,
which in turn promotesche a sua volta promuove la prolifera
tion and autophagy in AML cellszione e l'autofagia nelle cellule AML [150 ] [65].
P53
Altera
tzion
s of the tumor sui del gene oncosoppressor
genee TP53 asi r
e found in approximatelyiscontrano in circa il 5-15%
of AML cases and dei casi di AML e, frequent
emente, nei pazienti più anziani [ 151 , 152 ]. È stato proposto che il
y ruolo in older patientsdell'autofagia nello sviluppo dell'AML possa essere determinato dallo stato [66][67].di It has been proposed that the role of autophagy in the development of AML may be determined by TP53 status.
For Per l'AML TP53 wild-type
AML, TP53,i r
ice
searchers demonstrated that phrcatori hanno dimostrato che il blocco farmacologic
al blockade of autophagy achieves tho dell'autofagia ottiene benefici terapeutic
benefits, whereas AMLs harboringi, mentre le AML che ospitano TP53 muta
tzion
si failTP53 tno
respond to autophagy inhibition by hydroxychloroquinen rispondono all'inibizione dell'autofagia da parte dell'idrossiclorochina (HCQ)
[ 153 , 154 [68][69]].
The L'us
e of autophagic inho di inibitor
s may be i autofagici può essere una poten
tial thziale strategia terapeutic
strategy to use, a da utilizzare, in partic
ularly for the treatment of wild-typeolare per il trattamento di TP53 AML
. For AML with di tipo selvaggio. Per l'AML con TP53 muta
tzion
si TP53 ,
le vie auto
phagic pathways may be a thfagiche possono essere un'opzione terapeutic
option to be used for the a da utilizzare per l'elimina
tion of thzione del mutante
T TP53 T mutant.
AUn
other study demonstrated that stimulation of altro studio ha dimostrato che la stimolazione della macroauto
phagy byfagia da parte di 17-AAG,
an HSP90 inhun inibitor
, results in the e HSP90, provoca la degrada
tion ofzione di TP53 R248Q
in AML cells and also enhances the transcription of autophagy-nelle cellule AML e migliora anche la trascrizione dei geni associat
ed genesi all'autofagia [155 ] [70].
FuInoltr
thermore,e, le prove accumulat
ing evidencee indica
tes thatno che TP53 a
ctivated by ttivato da una variet
y ofà di stress cellular
stresses can trigger autophagy through thei può innescare l'autofagia attraverso la transa
ctivation ofttivazione di geni pro-auto
phagic genes, includingfagici, tra cui DRAM1 (DNA (mod
amage regulat
ed autophagy modulatorore autofagico regolato dal danno al DNA 1),
SESN1 (sestrin 1)
, and ( e (sestrin
SESN2 )
[ 155 [70][71][72][73], 156 , 157 , 158 ].
AUno studio recent
study highlighted the role of autophagy in AML cells, in thee ha evidenziato il ruolo dell'autofagia nelle cellule AML, nel conte
xt of p53-sto dell'apoptosi mediat
ed apoptosis, which isa da p53, che è associat
ed with increased cytotoxicity to treatment with MDM2 inha ad una maggiore citotossicità al trattamento con inibitor
s andi MDM2 e Ara-C
when quando il miR-10a
is inhibitedè inibito [159 ] [74].
ThLa strate
gia antileu
kemic strategy based on the use ofcemica basata sull'uso di inibitori MDM2/X
inhibitors innei tumori p53 wild-type p
53 tumors to restore the normal and active conformation ofer ripristinare la conformazione normale e attiva di p53, MDM2
ande MDMX
has not been extensively testednon è stata ampiamente testata [160 ] [75].
ThPer
efore, the use of tanto, l'uso di una combina
tion of treatmentszione di trattamenti, inclu
ding MDM2 inhsi gli inibitor
s with autophagici MDM2 con modulator
s, could be a noveli autofagici, potrebbe essere una nuova strateg
y to improve the treatment ofia per migliorare il trattamento dell'AML p53 wild-type
p53 AML.
PhI trattamenti farmacologic
al treatments that i che modula
te autophagy in AML patients carryingno l'autofagia nei pazienti con LMA portatori p53di muta
tzion
si p53 part
iecipa
te in theno alla degrada
tion ofzione delle proteine p53 aberrant
p53 proteins. Point mutation ofi. La mutazione puntiforme di TP53 atnel theresiduo amino
acid residueacidico R428 (R248Q),
with gain-of-function activity, gives rise to malignant activity in lung cancer cellscon attività di guadagno di funzione, dà origine ad attività maligna nelle cellule tumorali del polmone [ 161 ] [76]e una
nd a loss of tumor su perdita della funzione di soppress
or function in AMLione tumorale nell'AML [ [77]162 ].
IÈ interes
tingly, treatment with thsante notare che il trattamento con l'inibitore Hsp90
inhibitor (17-AAG)
results in the activation of chaperone-determina l'attivazione dell'autofagia mediat
ed autophagy, whicha da chaperone, che induce
s the la degrada
tion of thezione della proteina aberrant
proteine p53R248Q
in AML cells. Notably, undernelle cellule AML. In particolare, in condi
tions of zioni di stress metabolic
stresso, 17-AAG induce
s the l'intera
ction betweenzione tra p53R248Q
and the e la proteina chaperone
protein Hsc70,
triggering chaperone-innescando l'autofagia mediat
ed autophagy to a da chaperone per degrad
are p53R248Q
[70][ 155 ].
ThQues
e data open newti dati aprono nuove opportunit
ies for future studies that could elucidate the functional involvement of different types of autophagy and theirà per studi futuri che potrebbero chiarire il coinvolgimento funzionale di diversi tipi di autofagia e la loro conne
ction with thessione con i meccanismi molec
ular mechanisms for improving anticancer tholari per migliorare le terapie
s against AML harboring the different TP53 antitumorali contro l'AML che ospita le diverse variant
si TP53 .
IDH1/2 (isocitrate dehydrogenase)
RI recent
advances ini progressi nella bioinformatic
s have enabled the a hanno consentito l'identifica
tion of severalzione di diverse mutazioni epigenetic
mutations affecting AML, includinghe che interessano l'AML, tra cui IDH1/2 , Tet met
hylcytosine dioxygenaseilcitosina diossigenasi 2 (
TET2 ), DNA met
hyiltransferas
ei 3A (
DNMT3A )
ande ASXL1 ,
all of which artutte associate
d with the path alla patogenesi
s of AML dell'AML [ 163 , 164 , [78][79][80]165].
IDHLe protein
s are e IDH sono isocitrat
e dehyo deidrogenas
esi, implicate
d in vari
ous processi biologic
al processes, such asi, come il metabolismo energ
y metabolism, histetico, la demetilazione de
methylation, DNAll'istone, la modifica
tion, and adaptation to hypoxia. Furtherzione del DNA e l'adattamento all'ipossia. Sono necessari ulteriori studi
es are needed to investigat per studiare terapie innovative
therapies based on targeted autophagybasate sull'autofagia mirata in combina
tion with DNA hypomethylation to treat AMLs harboring certain types ofzione con l'ipometilazione del DNA per trattare le AML che ospitano determinati tipi di alterazioni epigenetic
alhe.
Muterations.
DNMT3A
Mutations in the zioni nel gene DNMT3A gene,
an enzyme involved in CpGun enzima coinvolto nella metilazione dei dinucleotid
e methylation, are i CpG, sono present
ini nel 20-23%
of adult patients withdei pazienti adulti con AML de novo
AML[ 165 [80]].
SeDiver
alsi studi
es have shown that treatment of AML patients with hanno dimostrato che il trattamento di pazienti affetti da LMA con agenti che inibiscono la DNA met
hyiltransferas
e-inhibiting agents, such as ai, come l'azacitidin
ea (5-aza-2′-deo
xycytidinessicitidina), induce
s autophagic activity in AML leukemia cells l'attività autofagica nelle cellule di leucemia AML [166 ] [81].
AUno stud
y in a DNMT3A R878H io cond
iott
ional o su un modello murino knock-in
mouscondizionale
modelDNMT3A R878H, u
sed tilizzato p
redict the er prevedere gli specific
long noncoding RNAs i RNA lunghi non codificanti (lncRNA
s) regulated by) regolati dalle mutazioni DNMT3A mutation
s in AML, firstell'AML, ha prima identifi
edcato 23
lncRNAs espressi in modo differen
tzia
lly expressed, then downstream target genes regulated by theseto , quindi i geni bersaglio a valle regolati da questi lncRNA
s , inclu
dingso ATP6V1A,
aun gene criti
cal autophagy-related gene, overexco correlato all'autofagia, la cui sovraespression
of which is e è associat
ed with poor pra a prognosi
s in infausta nell'AML
[82][ 167 ].
However, there is still little evidence for a direct involvement of DNMT3ATuttavia, ci sono ancora poche prove di un coinvolg
ene mutationsimento diretto delle mutazioni del gene DNMT3A con wattivit
h autophagic activity in à autofagica nell'AML.
FSono necessari urtherlteriori studies are needed to understand the functiona per comprendere il significance of autophagyto funzionale dell'autofagia associated with different gene mutations in AML cellsa a diverse mutazioni genetiche nelle cellule AML.