Adductomics to transformacyjne narzędzie badań biomedycznych, które wykorzystuje podejście „omiczne” do charakteryzowania i ilościowego określania egzogennych i endogennych reaktywnych związków, na które narażona jest jednostka; wykorzystanie biomarkerów adduktów specyficznych dla związków. Narażenie na chemikalia jest na ogół napędzane przez różne czynniki, takie jak środowisko, genetyka i styl życia, charakteryzujące się wysokim poziomem zmienności międzyosobowej i zawiera element życia, co czyni go unikalnym dla każdej osoby.
- adductomics
- DNA adduct
- protein adduct
- cancer detection tool
1. Wstęp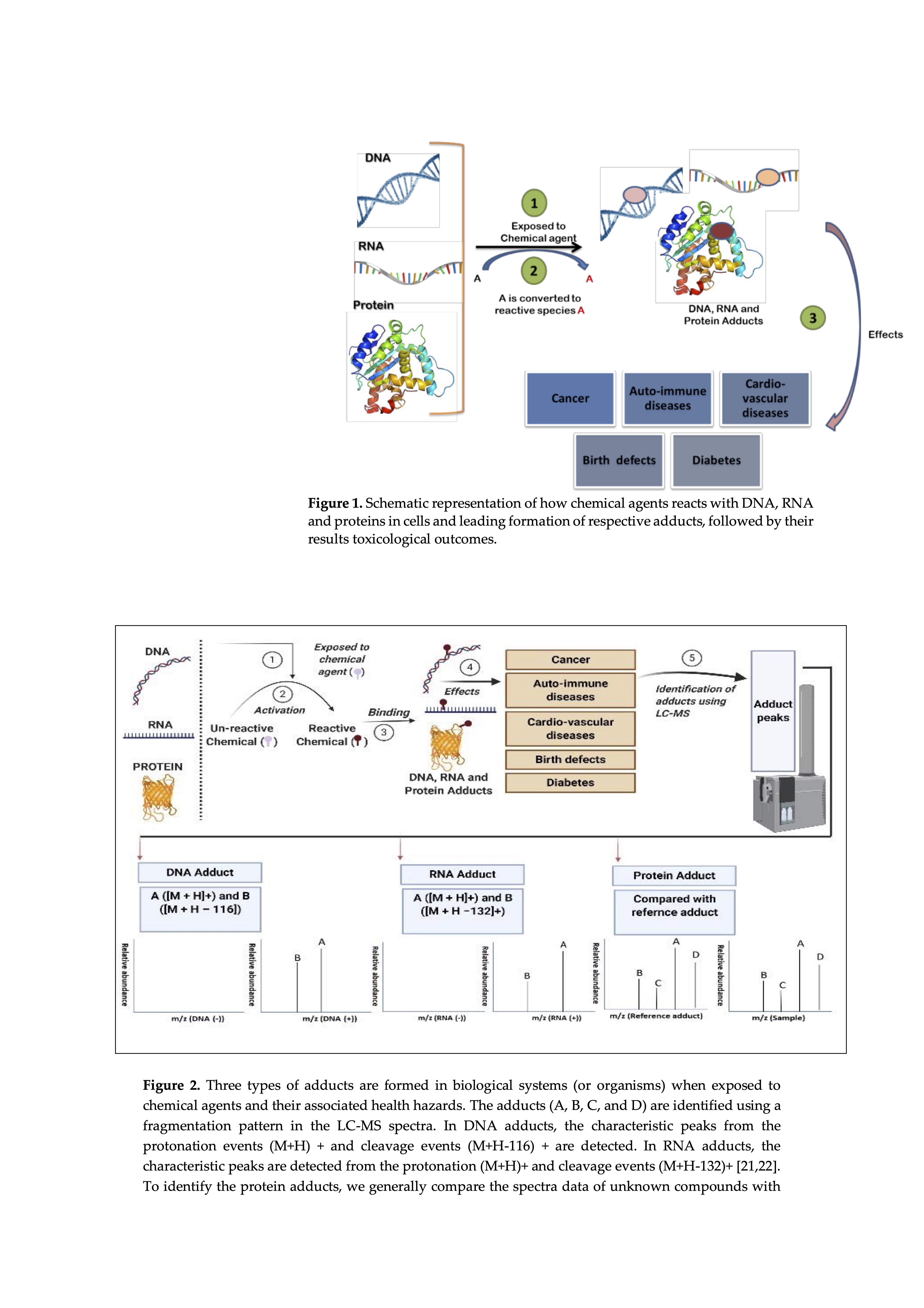
Adduktomika stała się najbardziej znaczącą techniką w ostatnich czasach i jest jedną z szybko rozwijających się dyscyplin, która może radykalnie zmienić krajobraz badań toksykologicznych. Prawdopodobnie nadszedł czas, aby adduktomicy dołączyli do elitarnego klubu słów takich jak genomika, proteomika i metabolomika. Chociaż termin adduktomika pojawił się po raz pierwszy w czasopiśmie opublikowanym w 2006 roku [ 1]] i obecnie jego zastosowania sięgają niemal wszystkich obszarów badań toksykologicznych. Adductomics to transformacyjne narzędzie badań biomedycznych, które wykorzystuje podejście „omiczne” do charakteryzowania i ilościowego określania egzogennych i endogennych reaktywnych związków, na które narażona jest jednostka; wykorzystanie biomarkerów adduktów specyficznych dla związków. Narażenie na chemikalia jest na ogół napędzane przez różne czynniki, takie jak środowisko, genetyka i styl życia, charakteryzujące się wysokim poziomem zmienności międzyosobowej i zawiera element życia, co czyni go unikalnym dla każdej osoby.
Adduktomika koncentruje się głównie na badaniu adduktów powstałych w wyniku modyfikacji kowalencyjnych, które są w przyrodzie nieodwracalne z bio-makrocząsteczkami przez egzogenne lub endogenne reaktywne związki elektrofilowe. Związki reaktywne oddziałują z nukleofilowymi gorącymi punktami (miejscami podatnymi na elektrofile) obecnymi w DNA, lipidach, białkach, RNA i innych makrocząsteczkach, prowadząc do tworzenia adduktów (jak pokazano na Rysunku 1). Biomonitorowanie reaktywnych metabolitów niezależnie od ich pochodzenia, czy to egzogennych, czy endogennych, stanowi wyzwanie ze względu na ich krótki czas życia in vivo, a adduktomika zapewniła unikalną możliwość wykrycia kowalencyjnych adduktów, które są stosunkowo stabilne i długowieczne. Badania nad adduktomią szeroko wykorzystują dwa podejścia (1) ukierunkowane i (2) nieukierunkowane; Pierwsza metoda skupia się na wykrywaniu konkretnych adduktów po ekspozycji na określone czynniki chemiczne, a późniejsza kategoria ma na celu scharakteryzowanie wszystkich adduktów poprzez wiązania kowalencyjne [ 2 , 3 ].
W niektórych przypadkach środki chemiczne w postaci naturalnej nie wiążą się bezpośrednio z cząsteczkami biologicznymi, tworząc addukty, jednak transformacja do reaktywnych metabolitów zachodzi przez enzymy metaboliczne, takie jak układy cytochromu P450, a utworzone reaktywne metabolity mogą wiązać się z białkiem, RNA i DNA [ 4]. , 5 ]. Reaktywne elektrofile generowane z kowalencyjnych wiązań genotoksycznych z DNA mogą zachodzić poprzez kilka mechanizmów: (1) aryloaminacja [ 6 ], (2) alkilacja [ 7 ], (3) rozwój sieciowania bis-elektrofilowego [ 8 ] i (4) addukty z wysoce reaktywne związki pośrednie wytwarzane w wyniku peroksydacji lipidów [ 9 , 10 , 11] lub reaktywne formy tlenu. Typ i charakter utworzonych adduktów DNA jest zazwyczaj zależny od wielu czynników, takich jak struktura chemiczna reaktywnych substancji chemicznych, zdolność substancji chemicznej do interkalacji z DNA i charakter elektrofili. Dowody na obecność aflatoksyny, nitrozoaminy specyficznej dla tytoniu (NNK), wielopierścieniowych węglowodorów aromatycznych (B[a]P), heterocyklicznej aminy aromatycznej i innych toksyn egzogennych tworzą inny typ adduktów DNA ze względu na ich różne właściwości strukturalne i chemiczne [ 12 ]. Tworzenie adduktów może mieć znaczący wpływ na system biologiczny i prowadzić do szkodliwych powikłań zdrowotnych [ 13 ], takich jak cukrzyca, choroby neurodegeneracyjne, choroby autoimmunologiczne, nowotwory, wady wrodzone [ 14 ] i choroby sercowo-naczyniowe [15 ]. Zrozumienie adduktów indukowanych chemicznie za pomocą adduktomiki może być niezbędne do uzyskania lepszego wglądu w te choroby i dostarczenia nowych informacji na temat skutecznego opracowywania leków. Adduktomika może mieć różnorodne zastosowania praktyczne w różnych dziedzinach, a jej rola jest oczywista (1) prognozowanie chorób, (2) ocena stanu zdrowia środowiska [ 14 ], (3) rozwój medycyny spersonalizowanej i precyzyjnej [ 16 ], (4) wykrywanie biomarkerów dla różnych ekspozycji chemicznych [ 17 , 18 , 19]. Pomiar adduktów DNA powstałych w wyniku ekspozycji na potencjalny czynnik rakotwórczy w narządach docelowych jest jedną z podstawowych metod oceny zdolności genotoksycznej związku chemicznego i służy jako najbardziej zaawansowana metoda określania potencjału genotoksyczności substancji chemicznej. Poza tym, adduktomika identyfikuje również leżące u podstaw czynniki ryzyka patogenezy i leżące u podstaw molekularne mechanizmy toksyczności indukowanej chemicznie. Dane z adduktomiki służyłyby również jako przewodnik dla agencji regulacyjnych i umożliwiały innym zainteresowanym stronom podejmowanie środków zapobiegawczych przeciwko narażeniu na toksyczną substancję chemiczną.
Szybkie doskonalenie metod i narzędzi w identyfikacji i ilościowej ocenie adduktów sprawiło, że adduktomika stała się jedną z najbardziej obiecujących dyscyplin toksykologicznych. Pobieranie próbek tkanek i przygotowanie próbek jest jednym z kluczowych czynników w wykrywaniu różnych adduktów. Ostatnie postępy naukowe oraz zwiększona precyzja metod wykrywania i przygotowania próbek pozwalają na ich nieinwazyjne pobieranie próbek i wykorzystanie płynów ustrojowych (osocze krwi lub surowica, mocz) tzw. biopsję płynną. Stosowanie nieinwazyjnych metod, takich jak pobieranie próbek płynnych, zapewnia różne korzyści, takie jak 1) próbki mogą być pobierane w różnych odstępach czasu bez powodowania dużego dyskomfortu u pacjentów 2) łatwość pobierania i przechowywania 3) łatwość transportu.20 ]. Spośród narzędzi szeroko stosowaną i odpowiednią metodą oceny jakościowego i ilościowego tworzenia adduktów, w tym identyfikacji kowalencyjnych miejsc koniugatu w bionukleofilach, jest wysokorozdzielcza spektrometria mas (HRMS). Wzór fragmentacji w spektrometrii mas jest wykorzystywany do identyfikacji różnych typów adduktów, a godnym uwagi aspektem dotyczącym adduktów DNA i RNA jest prawie powszechna utrata rybozy i dezoksyrybozy z macierzystej cząsteczki dająca charakterystyczne piki przy (M + H-116)+ i ( M + H−132)+ odpowiednio [ 20 , 21 ]. Z drugiej strony, nieznane addukty białkowe identyfikuje się przez porównanie danych spektralnych testowanych adduktów z adduktem referencyjnym ( Rysunek 2). Po pierwsze, addukty odniesienia powinny zostać zsyntetyzowane przy założeniu konkretnego elektrofila, a następnie zostaną dopasowane do nowych adduktów będących przedmiotem dalszych badań. Przez dodanie proponowanych prekursorów elektrofilowych do osocza lub pełnej krwi/lizatu można wytworzyć addukty odniesienia i poddać je fragmentacji przy użyciu LC-MS. Syntetyczne addukty będą następnie dalej porównywane z nowymi lub niezidentyfikowanymi adduktami z m/z jonów prekursorowych, badając również wzorce fragmentacji i czasy retencji. Co więcej, podejście to przyczynia się również do generowania obszernej bazy danych adduktów białek referencyjnych, dzięki czemu identyfikacja nieznanych adduktów białkowych staje się znacznie łatwiejsza [ 22 ].
2. Zastosowanie adduktomiki
Adduktomika znajduje zastosowanie w ocenie zanieczyszczeń i dostarcza informacji dotyczących ich toksycznego wpływu na systemy biologiczne sygnalizujące stan zdrowia środowiska. Na przykład wielopierścieniowe węglowodory aromatyczne (WWA), toksyczne zanieczyszczenia, to grupa podobnych strukturalnie węglowodorów uwalnianych do atmosfery w wyniku niecałkowitego spalania materii organicznej, dymu tytoniowego, zanieczyszczenia powietrza w miastach oraz emisji spalin samochodowych [ 29 ]. WWA mogą tworzyć addukty z DNA poprzez reaktywne związki pośrednie, gdy są aktywowane za pomocą systemów cytochromu P-450, co czyni je wysoce rakotwórczymi [ 30].]. Jedną z takich reaktywnych form elektrofilowych tworzonych przez CYP 1A1 i CYP 1B1 jest epoksyd PAH-dihydro-diolu, który może reagować z grupami egzocyklicznymi obecnymi w nukleotydach, takimi jak guanina, adenina i cytozyna obecnymi w DNA [ 31 ]. Podobnie, wiele adduktów PAH-DNA tworzy się z innymi reaktywnymi półproduktami u osób narażonych na PAH, a utworzone addukty DNA bada się za pomocą znakowania 32P-Post i LC-MS [ 30 ]. Jednoczesna ocena całej puli adduktów PAH-DNA u osób wyposaża nas w kompleksowy profil ekspozycji i ułatwia lepsze zrozumienie podstawowych szlaków mechanistycznych [ 32].]. W innym badaniu ustalono związek między powstawaniem adduktów WWA-DNA przy zanieczyszczeniu powietrza atmosferycznego u narażonych matek i noworodków w Polsce, co widać na krzywej dawka-odpowiedź, która wykazała proporcjonalny wzrost liczby adduktów DNA wraz ze stopniem zanieczyszczenia powietrza [ 33 ]. W populacji śródziemnomorskiej addukty o dużej objętości są skorelowane z zanieczyszczeniem środowiska ozonem, które przyczynia się do powstawania smogu fotochemicznego [ 34 ]. Hylland i in. [ 35] wykorzystali addukty DNA jako charakterystyczny biomarker do zbadania stopnia zanieczyszczenia w różnych miejscach w regionie północno-wschodniego Atlantyku w pobliżu wybrzeża i na morzu. Addukt jako biomarker (addukt DNA) ostrzega narażenie na ryzyko poprzez dostarczanie wczesnych informacji ostrzegawczych i pomaganie w poprawie oceny zagrożeń dla organizmów wodnych i oceny ryzyka ekologicznego [ 36]]. Ujawniono również, że addukty DNA (addukty PAH-DNA) pomogłyby również określić biologicznie skuteczną dawkę ekspozycji na PAH, informując o obecności i zakresie zanieczyszczenia środowiska oraz jego związku z rozwojem raka. WWA są wszechobecne, a ich obecność w mieszaninach ropy i gazu powoduje skażenie ekosystemu wodnego podczas poszukiwań ropy i gazu. Wykrywanie adduktów PAH-DNA może również służyć jako potencjalne biomarkery skażenia środowiska i badania genotoksyczności w organizmach wodnych [ 37 ]. Ponadto w kilku raportach przedstawiono dowody na wpływ ropy naftowej i gazu produkcyjnego na tworzenie adduktów DNA w organizmach morskich zarówno u zwierząt laboratoryjnych, jak i in vivo po dużych wyciekach ropy naftowej [ 38 , 39 , 40].
Co więcej, wykrycie adduktów DNA indukowanych oksaplatyną u pacjentów z rakiem jelita grubego z FOLFOX (skojarzona terapia lekowa zawierająca kwas folinowy, fluorouracyl i oksaliplatyna) pomoże w zaprojektowaniu i zoptymalizowaniu lepszych strategii leczenia pacjentów z rakiem. Po leczeniu FOLFAX, wykryte addukty oksaplatyny-DNA w PBMC były proporcjonalne do redukcji guza, co czyni addukty Drug-DNA potencjalnym biomarkerem w leczeniu raka [ 50 ].
Addukty DNA to fizyczne kompleksy utworzone z DNA w wyniku interakcji reaktywnych form chemicznych z DNA, a wykrycie tych adduktów posłużyłoby jako potencjalne markery do określania „biologicznie skutecznej dawki” dla obecności substancji rakotwórczych w dymie tytoniowym i może pomóc w lepszym monitorowaniu zdrowia palaczy. Kilka badań wykazało, że ekspozycja na dym tytoniowy może potencjalnie indukować tworzenie adduktów DNA w badaniach in vivo i wykazały pozytywną korelację z karcynogenezą. Ponadto wykrywanie adduktów DNA może również zapewnić kompleksowy pomiar narażenia na czynniki rakotwórcze, również w ocenie i przewidywaniu ryzyka raka. Kilka badań klinicznych i epidemiologicznych ustaliło związek między zwiększonym poziomem adduktów DNA a prawdopodobieństwem wystąpienia nowotworów związanych z tytoniem, takich jak rak płuc, głowy, szyi i pęcherza moczowego [52 , 53]. Podczas gdy profil adduktów DNA zapewnia obrazy ekspozycji, obciążenie adduktami DNA ocenia ryzyko kancerogenezy. Uważa się również, że indukcja adduktów DNA w limfocytach krwi jest związana z rozwojem raka głowy i szyi. Jednak zależność dawka-odpowiedź między paleniem a adduktami DNA w eksponowanych narządach nie jest w pełni scharakteryzowana i w rzeczywistości związek ten jest skomplikowany z powodu niespójności w badaniach epidemiologicznych i zakłada się polimorfizm genetyczny (w metabolizmie czynników rakotwórczych (np. GSTP1) i naprawa DNA (np. XRCC1)) jest podstawową przyczyną. W przypadkach guzów we wczesnym stadium kancerogenezy tytoniu, indukowanych przez mutacje p53 i addukty DNA, zaobserwowano, że poziomy adduktów DNA były skorelowane ze zmianami somatycznymi (np. 3p21 LOH) [ 69 ].
Ocena toksyczności genetycznej ma wysoki priorytet w zarządzaniu ryzykiem bezpieczeństwa podczas opracowywania nowych związków chemicznych i czyni to poprzez ocenę rakotwórczości i mutagenności danej substancji chemicznej, pomagając w ten sposób w identyfikacji zagrożeń i charakterystyce ryzyka czynników chemicznych [ 97 , 98 ]. Tradycyjnie potencjały genotoksyczności, mutagenności i rakotwórczości substancji chemicznej ocenia się za pomocą testu Amesa, testu chromosomalnego i testu mikrojądrowego [ 99 , 100]. Jednak wyzwaniem związanym z tymi metodami in vitro jest wysoki odsetek wyników fałszywie dodatnich, które wymagają opracowania nowych metodologii i opartego na szlaku zrozumienia toksyczności, co może zapewnić dokładniejszy obraz uszkodzeń DNA, który może bezpośrednio wykryć DNA modyfikacje i uszkodzenia DNA na poziomie molekularnym [ 101 , 102 , 103 ]. Tutaj adduktomika DNA okazuje się potencjalnym kandydatem na metodologię, która mogłaby kompleksowo zbadać uszkodzenia DNA poprzez bezpośrednią detekcję molekularną poprzez identyfikację i kwantyfikację adduktów DNA [ 104 , 105 , 106]. Test mikrojądrowy jest jednym z szeroko stosowanych testów in vitro do oceny uszkodzeń DNA, ale obecnie jest uzupełniany przez adduktomię DNA, aby zniwelować błąd ze względu na fałszywie dodatnie wyniki testu, co oznacza potwierdzającą rolę adduktomiki DNA w innych ocena genotoksyczności in vitro [ 107 , 108 ].
3. Inne aplikacje
Oprócz powyższego, inne zastosowania adduktomiki przedstawiono w tabeli 1 poniżej wraz z tytułami publikacji wraz z nowatorskimi zastosowaniami adduktomiki:
4. Aktualne wyzwania i perspektywy na przyszłość
Postęp w narzędziach diagnostycznych i pojawienie się nowych technologii dały początek zastosowaniom adduktomiki. Jednak nadal istnieją wyzwania, którym należy sprostać, aby w pełni wykorzystać potencjał adduktomiki w ocenie toksykologicznej i środowiskowej chemikaliów. Chociaż metody pozyskiwania zależne od danych i niezależne od danych (w nieukierunkowanych „technologiach omikowych”) zostały opracowane w celu jednoczesnego badania wielu adduktów, należy zająć się przeszkodami w przetwarzaniu danych, aby uzyskać dokładny obraz substancji toksycznych [ 104].]. Niska częstotliwość adduktów DNA w puli próbek stanowi również poważne wyzwanie dla obecnego oprogramowania w realistycznej ocenie, które wykorzystuje powszechne metody pozyskiwania danych. To zapotrzebowanie na kontynuację oprogramowania do przetwarzania danych i ulepszenia algorytmów do wykrywania adduktów, nawet w niskich stężeniach, które są krytyczne dla zrozumienia patogenezy [ 143]]. Istnieje szereg ulepszeń w przygotowaniu próbek i oczyszczeniu, jeśli chodzi o wykrywanie adduktów hydrofilowych. Ponadto niepełna hydroliza enzymatyczna nie generuje i nie obserwuje pewnych typów adduktów DNA, co wymaga kompleksowej oceny zalet i wad kilku enzymów pod kątem hydrolizy DNA i ich optymalnego wykorzystania. W adduktach o masach cząsteczkowych poniżej 70 KDa istnieje niewiele struktur prawdopodobnych, a ich identyfikacja nie jest kłopotliwa, natomiast w adduktach o większych masach cząsteczkowych ich charakterystyka jest niezwykle trudna ze względu na poszerzone możliwości i amplifikowane permutacje; jest to kwestia niepokojąca, nawet jeśli moglibyśmy dokonać dokładnych pomiarów masy i wytworzyć widma fragmentacji jonów. Upośledzenie to można przezwyciężyć poprzez stworzenie bazy adduktów, która dostarczyłaby gotowych informacji na temat adduktów; niestety, nie ma specyficznej bazy danych dla adduktomiki, mimo że codziennie na całym świecie charakteryzowane są setki adduktów DNA, stworzenie takiej bazy wiąże się z dokładnym przeszukiwaniem literatury wzorów molekularnych już scharakteryzowanych adduktów. Widma fragmentacji wytworzone zarówno z pułapki jonowej, jak i fragmentacji typu kwadrupolowego na poziomach MS2 i MS3, wykazane przy różnych energiach zderzeń, byłyby przydatne, gdyby zostały skompilowane i zintegrowane z bazą danych. Obecnie bazy danych, takie jak Search for Species Data by Molecular Weight dostarczane przez NIST (National Institute of Standards and Technologies) [ nie ma specyficznej bazy danych dla adduktomiki, mimo że codziennie na całym świecie charakteryzowane są setki adduktów DNA, stworzenie takiej bazy wiąże się z dokładnym przeszukiwaniem literatury wzorów molekularnych już scharakteryzowanych adduktów. Widma fragmentacji wytworzone zarówno z pułapki jonowej, jak i fragmentacji typu kwadrupolowego na poziomach MS2 i MS3, wykazane przy różnych energiach zderzeń, byłyby przydatne, gdyby zostały skompilowane i zintegrowane z bazą danych. Obecnie bazy danych, takie jak Search for Species Data by Molecular Weight dostarczane przez NIST (National Institute of Standards and Technologies) [ nie ma specyficznej bazy danych dla adduktomiki, mimo że codziennie na całym świecie charakteryzowane są setki adduktów DNA, stworzenie takiej bazy wiąże się z dokładnym przeszukiwaniem literatury wzorów molekularnych już scharakteryzowanych adduktów. Widma fragmentacji wytworzone zarówno z pułapki jonowej, jak i fragmentacji typu kwadrupolowego na poziomach MS2 i MS3, wykazane przy różnych energiach zderzeń, byłyby przydatne, gdyby zostały skompilowane i zintegrowane z bazą danych. Obecnie bazy danych, takie jak Search for Species Data by Molecular Weight dostarczane przez NIST (National Institute of Standards and Technologies) [ Widma fragmentacji wytworzone zarówno z pułapki jonowej, jak i fragmentacji typu kwadrupolowego na poziomach MS2 i MS3, wykazane przy różnych energiach zderzeń, byłyby przydatne, gdyby zostały skompilowane i zintegrowane z bazą danych. Obecnie bazy danych, takie jak Search for Species Data by Molecular Weight dostarczane przez NIST (National Institute of Standards and Technologies) [ Widma fragmentacji wytworzone zarówno z pułapki jonowej, jak i fragmentacji typu kwadrupolowego na poziomach MS2 i MS3, wykazane przy różnych energiach zderzeń, byłyby przydatne, gdyby zostały skompilowane i zintegrowane z bazą danych. Obecnie bazy danych, takie jak Search for Species Data by Molecular Weight dostarczane przez NIST (National Institute of Standards and Technologies) [146 ], UNIMOD [ 147 ], Baza danych ludzkich metabolomów [ 142 ], Baza danych toksycznych ekspozycji [ 143 ], Baza danych Exposome-Explorer [ 144 ]] znaleźć zastosowanie w adduktomice. Jednak wspomniane powyżej bazy danych nie są specyficzne dla adduktomiki, co wymaga stworzenia dedykowanej bazy danych, która może ułatwić łatwą identyfikację nieznanych adduktów. Istnieje potrzeba opracowania bardziej solidnej i prostej technologii w celu dalszego doskonalenia pobierania próbek, jak sugerowano powyżej, bardziej skoncentrowane podejście do nieinwazyjnego pobierania próbek cieczy, wymagana jest optymalizacja metod przygotowania próbek, które mogą dawać precyzyjne i powtarzalne wyniki. Obecne techniki analityczne są bardzo czasochłonne i drogie w badaniu próbek, dalszy rozwój opłacalnych technik analitycznych może jeszcze bardziej wzmocnić zastosowania adduktomiki w badaniach biomedycznych.
References
- Kanaly, R.A.; Hanaoka, T.; Sugimura, H.; Toda, H.; Matsui, S.; Matsuda, T. Development of the Adductome Approach to Detect DNA Damage in Humans. Antioxidants Redox Signal. 2006, 8, 993–1001.
- Carlsson, H.; Rappaport, S.M.; Törnqvist, M. Protein Adductomics: Methodologies for Untargeted Screening of Adducts to Serum Albumin and Hemoglobin in Human Blood Samples. High-Throughput 2019, 8, 6.
- Preston, G.W.; Phillips, D.H. Protein Adductomics: Analytical Developments and Applications in Human Biomonitoring. Toxics 2019, 7, 29.
- Guengerich, F.P. Common and Uncommon Cytochrome P450 Reactions Related to Metabolism and Chemical Toxicity. Chem. Res. Toxicol. 2001, 14, 611–650.
- Rendic, S.; Guengerich, F.P. Contributions of Human Enzymes in Carcinogen Metabolism. Chem. Res. Toxicol. 2012, 25, 1316–1383.
- Beland, F.A.; Beranek, D.T.; Dooley, K.L.; Heflich, R.H.; Kadlubar, F.F. Arylamine-DNA adducts in vitro and in vivo: Their role in bacterial mutagenesis and urinary bladder carcinogenesis. Environ. Health Perspect. 1983, 49, 125–134.
- Shrivastav, N.; Li, D.; Essigmann, J.M. Chemical biology of mutagenesis and DNA repair: Cellular responses to DNA alkylation. Carcinogenesis 2009, 31, 59–70.
- Rajski, S.R.; Williams, R.M. DNA Cross-Linking Agents as Antitumor Drugs. Chem. Rev. 1998, 98, 2723–2796.
- Marnett, L.J. Lipid peroxidation—DNA damage by malondialdehyde. Mutat. Res. Mol. Mech. Mutagen. 1999, 424, 83–95.
- Tudek, B.; Zdżalik-Bielecka, D.; Tudek, A.; Kosicki, K.; Fabisiewicz, A.; Speina, E. Lipid peroxidation in face of DNA damage, DNA repair and other cellular processes. Free. Radic. Biol. Med. 2017, 107, 77–89.
- Marnett, L.J. Oxy radicals, lipid peroxidation and DNA damage. Toxicology 2002, 181, 219–222.
- Guo, J.; Turesky, R.J. Emerging Technologies in Mass Spectrometry-Based DNA Adductomics. High-Throughput 2019, 8, 13.
- Colombo, G.; Clerici, M.; Giustarini, D.; Rossi, R.; Milzani, A.D.G.; Dalle-Donne, I. Redox Albuminomics: Oxidized Albumin in Human Diseases. Antioxid. Redox Signal. 2012, 17, 1515–1527.
- Gorokhova, E.; Martella, G.; Motwani, N.H.; Tretyakova, N.Y.; Sundelin, B.; Motwani, H.V. DNA epigenetic marks are linked to embryo aberrations in amphipods. Sci. Rep. 2020, 10, 1–11.
- Zhang, H.; Ge, Y. Comprehensive Analysis of Protein Modifications by Top-Down Mass Spectrometry. Circ. Cardiovasc. Genet. 2011, 4, 711.
- Stornetta, A.; Zimmermann, M.; Cimino, G.D.; Henderson, P.T.; Sturla, S.J. DNA Adducts from Anticancer Drugs as Candidate Predictive Markers for Precision Medicine. Chem. Res. Toxicol. 2017, 30, 388–409.
- Harris, C.C. Future directions in the use of DNA adducts as internal dosimeters for monitoring human exposure to environmental mutagens and carcinogens. Environ. Heal. Perspect. 1985, 62, 185–191.
- La, D.K.; Swenberg, J.A. DNA adducts: Biological markers of exposure and potential applications to risk assessment. Mutat. Res. Genet. Toxicol. 1996, 365, 129–146.
- Jarabek, A.M.; Pottenger, L.H.; Andrews, L.S.; Casciano, D.; Embry, M.R.; Kim, J.H.; Preston, R.J.; Reddy, M.V.; Schoeny, R.; Shuker, D.; et al. Creating context for the use of DNA adduct data in cancer risk assessment: I. Data organization. Crit. Rev. Toxicol. 2009, 39, 659–678.
- Balbo, S.; Turesky, R.J.; Villalta, P.W. DNA Adductomics. Chem. Res. Toxicol. 2014, 27, 356–366.
- Takeshita, T.; Kanaly, R.A. In vitro DNA/RNA Adductomics to Confirm DNA Damage Caused by Benzopyrene in the Hep G2 Cell Line. Front. Chem. 2019, 7, 7.
- Rappaport, S.M. Genetic Factors Are Not the Major Causes of Chronic Diseases. PLOS ONE 2016, 11, e0154387.
- Grimmer, V.G. Environmental Carcinogens: Polycyclic Aromatic Hydrocarbons; CRC Press: Boca Raton, FL, USA, 1983.
- Singh, R.; Teichert, F.; Seidel, A.; Roach, J.; Cordell, R.; Cheng, M.-K.; Frank, H.; Steward, W.P.; Manson, M.M.; Farmer, P.B. Development of a targeted adductomic method for the determination of polycyclic aromatic hydrocarbon DNA adducts using online column-switching liquid chromatography/tandem mass spectrometry. Rapid Commun. Mass Spectrom. 2010, 24, 2329–2340.
- Wang, J.J.; Marshall, W.D.; Frazer, D.G.; Law, B.; Lewis, D.M. Characterization of DNA adducts from lung tissue of asphalt fume-exposed mice by nanoflow liquid chromatography quadrupole time-of-flight mass spectrometry. Anal. Biochem. 2003, 322, 79–88.
- Ewa, B.; Danuta, M.-. Švach Polycyclic aromatic hydrocarbons and PAH-related DNA adducts. J. Appl. Genet. 2017, 58, 321–330.
- Whyatt, R.M.; Santella, R.M.; Jedrychowski, W.; Garte, S.J.; Bell, D.; Ottman, R.; Gladek-Yarborough, A.; Cosma, G.; Young, T.L.; Cooper, T.B.; et al. Relationship between ambient air pollution and DNA damage in Polish mothers and newborns. Environ. Heal. Perspect. 1998, 106, 821–826.
- Palli, D.; Saieva, C.; Grechi, D.; Masala, G.; Zanna, I.; Barbaro, A.; Decarli, A.; Munnia, A.; Peluso, M. DNA bulky adducts in a Mediterranean population correlate with environmental ozone concentration, an indicator of photochemical smog. Int. J. Cancer 2004, 109, 17–23.
- Hylland, K.; Skei, B.B.; Brunborg, G.; Lang, T.; Gubbins, M.J.; le Goff, J.; Burgeot, T. DNA damage in dab (Limanda limanda) and haddock (Melanogrammus aeglefinus) from European seas. Mar. Environ. Res. 2017, 124, 54–60.
- A Hagger, J.; Jones, M.B.; Leonard, P.; Owen, R.; Galloway, T.S. Biomarkers and integrated environmental risk assessment: Are there more questions than answers? Integr. Environ. Assess. Manag. 2006, 2, 312–329.
- Pampanin, D.M.; Brooks, S.J.; Grøsvik, B.E.; Le Goff, J.; Meier, S.; Sydnes, M.O. DNA adducts in marine fish as biological marker of genotoxicity in environmental monitoring: The way forward. Mar. Environ. Res. 2017, 125, 49–62.
- Lyons, B.; Stewart, C.; Kirby, M. The detection of biomarkers of genotoxin exposure in the European flounder (Platichthys flesus) collected from the River Tyne Estuary. Mutat. Res. Toxicol. Environ. Mutagen. 1999, 446, 111–119.
- Aas, E.; Baussant, T.; Balk, L.; Liewenborg, B.; Andersen, O.K. PAH metabolites in bile, cytochrome P4501A and DNA adducts as environmental risk parameters for chronic oil exposure: A laboratory experiment with Atlantic cod. Aquat. Toxicol. 2000, 51, 241–258.
- Harvey, J.; Lyons, B.; Page, T.; Stewart, C.; Parry, J. An assessment of the genotoxic impact of the Sea Empress oil spill by the measurement of DNA adduct levels in selected invertebrate and vertebrate species. Mutat. Res. Toxicol. Environ. Mutagen. 1999, 441, 103–114.
- Zimmermann, M.; Li, T.; Semrad, T.J.; Wu, C.-Y.; Yu, A.; Cimino, G.; Malfatti, M.; Haack, K.; Turteltaub, K.W.; Pan, C.-X.; et al. Oxaliplatin–DNA Adducts as Predictive Biomarkers of FOLFOX Response in Colorectal Cancer: A Potential Treatment Optimization Strategy. Mol. Cancer Ther. 2020, 19, 1070–1079.
- Johnson, L.A.; Malayappan, B.; Tretyakova, N.; Campbell, C.; MacMillan, M.; Wagner, J.E.; Jacobson, P.A. Formation of cyclophosphamide specific DNA adducts in hematological diseases. Pediatr. Blood Cancer 2012, 58, 708–714.
- Stornetta, A.; Villalta, P.W.; Gossner, F.; Wilson, W.R.; Balbo, S.; Sturla, S.J. DNA Adduct Profiles Predict in Vitro Cell Viability after Treatment with the Experimental Anticancer Prodrug PR104A. Chem. Res. Toxicol. 2017, 30, 830–839.
- National Toxicology Program (NTP). Toxicology and Carcinogenesis Studies of Furan (CAS NO. 110-00-9) in F344/N Rats and B6C3F1 Mice (Gavage Studies); NTP TR 402 (NIH Publication No. 93-2857); U.S. Department of Health and Human Services, Public Health: Washington, DC, USA, 1993; pp. 1–287.
- Löf, M.; Sundelin, B.; Liewenborg, B.; Bandh, C.; Broeg, K.; Schatz, S.; Gorokhova, E. Biomarker-enhanced assessment of reproductive disorders in Monoporeia affinis exposed to contaminated sediment in the Baltic Sea. Ecol. Indic. 2016, 63, 187–195.
- Löf, M.F.; Sundelin, B.; Bandh, C.; Gorokhova, E. Embryo aberrations in the amphipod Monoporeia affinis as indicators of toxic pollutants in sediments: A field evaluation. Ecol. Indic. 2016, 60, 18–30.
- Cimino, M.C. Comparative overview of current international strategies and guidelines for genetic toxicology testing for regulatory purposes. Environ. Mol. Mutagen. 2006, 47, 362–390.
- Thybaud, V.; Lorge, E.; Levy, D.; van Benthem, J.; Douglas, G.R.; Marchetti, F.; Moore, M.M.; Schoeny, R. Main issues addressed in the 2014-2015 revisions to the OECD Genetic Toxicology Test Guidelines. Environ. Mol. Mutagen. 2017, 58, 284–295.
- Kirkland, D.; Aardema, M.; Henderson, L.; Müller, L. Evaluation of the ability of a battery of three in vitro genotoxicity tests to discriminate rodent carcinogens and non-carcinogens: I. Sensitivity, specificity and relative predictivity. Mutat. Res. Toxicol. Environ. Mutagen. 2005, 584, 1–256.
- Hayashi, M.; Honma, M.; Takahashi, M.; Horibe, A.; Tanaka, J.; Tsuchiya, M.; Morita, T. Identification and Evaluation of Potentially Genotoxic Agricultural and Food-related Chemicals. Food Saf. 2013, 1, 2013003.
- Kirkland, D.; Pfuhler, S.; Tweats, D.; Aardema, M.; Corvi, R.; Darroudi, F.; Elhajouji, A.; Glatt, H.; Hastwell, P.; Hayashi, M.; et al. How to reduce false positive results when undertaking in vitro genotoxicity testing and thus avoid unnecessary follow-up animal tests: Report of an ECVAM Workshop. Mutat. Res. Toxicol. Environ. Mutagen. 2007, 628, 31–55.
- Sobus, J.R.; Wambaugh, J.; Isaacs, K.; Williams, A.; McEachran, A.; Richard, A.M.; Grulke, C.; Ulrich, E.; Rager, J.E.; Strynar, M.; et al. Integrating tools for non-targeted analysis research and chemical safety evaluations at the US EPA. J. Expo. Sci. Environ. Epidemiology 2018, 28, 411–426.
- Kanaly, R.A.; Matsui, S.; Hanaoka, T.; Matsuda, T. Application of the adductome approach to assess intertissue DNA damage variations in human lung and esophagus. Mutat. Res. Mol. Mech. Mutagen. 2007, 625, 83–93.
- Kato, K.; Yamamura, E.; Kawanishi, M.; Yagi, T.; Matsuda, T.; Sugiyama, A.; Uno, Y. Application of the DNA adductome approach to assess the DNA-damaging capability of in vitro micronucleus test-positive compounds. Mutat. Res. Toxicol. Environ. Mutagen. 2011, 721, 21–26.
- Rappaport, S.M.; Smith, M.T. Environment and Disease Risks. Science 2010, 330, 460–461.
- HEP. The Human Exposome Project: A Toolbox for Assessing and Addressing the Impact of Environment on Health. Available online: https://cordis.europa.eu/programme/id/H2020_SC1-BHC-28-2019 (accessed on 10 July 2021).
- Toxic Exposome Database, T3DB. Available online: http://www.t3db.ca/ (accessed on 10 July 2021).
- Molecular Weight Search. Available online: http://webbook.nist.gov/chemistry/mw-ser.html (accessed on 10 July 2021).
- Unimod. Available online: http://www.unimod.org/modifications_list.php (accessed on 10 July 2021).
- Human Metabolome Database. Available online: https://hmdb.ca/ (accessed on 17 September 2021).
- Neveu, V.; Moussy, A.; Rouaix, H.; Wedekind, R.; Pon, A.; Knox, C.; Wishart, D.S.; Scalbert, A. Exposome-Explorer: A manually-curated database on biomarkers of exposure to dietary and environmental factors. Nucleic Acids Res. 2017, 45, D979–D984.
